-
المساهمات
1068 -
تاريخ الانضمام
-
تاريخ آخر زيارة
-
عدد الأيام التي تصدر بها
43
نوع المحتوى
ريادة الأعمال
البرمجة
التصميم
DevOps
التسويق والمبيعات
العمل الحر
البرامج والتطبيقات
آخر التحديثات
قصص نجاح
أسئلة وأجوبة
كتب
دورات
كل منشورات العضو Ali Haidar Ahmad
-
هذه الطريقة نادرة الاستخدام عموماً. وهي أحد طرق تقسيم الداتا مثل StratifiedKFold ولكن يختلف عنه بأنه يقوم بأخذ نسبه متساوية من كل صنف في كل مجموعة.. في StratifiedKFold لو كان لدينا أربع عينات وكل منها يحمل 0و0و1و1 فمن الممكن أن يأخذ 0و0 والمجموعه الثانيه 1و1 وهذا تساوي أما Repeated Stratified KFold سوف يأخذ 0و1 في الأولى و1و0 في الثانية لكي تكون النسبة متساوية لكل صنف في كل مجموعة. يتم استخدامه عبر الموديل sklearn.model_selection. #استدعاء المكتبات from sklearn.model_selection import RepeatedStratifiedKFold قمنا باستدعاء المكتبة numpy لتشكيل الداتا واستدعاء الوظيفه RepeatedStratifiedKFold من الوحدة model_selection في مكتبة sklearn #تشكيل الداتا الدخل والخرج X = np.array([[1, 2], [3, 4], [5, 6], [7, 8]]) y = np.array([1, 0, 1, 0]) حيث X مصفوفة أبعادها 4 أسطر وعمودين وy عمود واحد بأربع أسطر #RepeatedStratifiedKFold rskf = RepeatedStratifiedKFold(n_splits=5,n_repeats=10, random_state=None) الوسيط الأول n_splits عدد صحيح لتحديد عدد الأجزاء folds وهي افتراضية في sklearn من 3 إلى 5. الوسيط الثاني n_repeats وهو نقطة الاختلاف عن StratifiedKFold هو عدد صحيح يمثل عدد KFolds. الوسيط الثالث random_state للتحكم بآلية التقسيم. نفس الأمر في StratifiedKFold مرور حلقة حيث يتم استخدام اثنين من الاندكسات في الحلقة للمرور على التدريب والاختبار وتحوي rskf.split(X, y) هذه الاندكسات . نقوم بطباعة الاندكس للتدريب والاختبار في كل محاولة وبعدها تخزين التدريب والاختبار ومن ثم طباعة كل منها: for train_index, test_index in rskf.split(X, y): print("TRAIN:", train_index, "TEST:", test_index) X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index] print('X_train:\n '+str(X_train),end='\n\n') print('X_test:\n '+str(X_train),end='\n\n') print('y_train:\n '+str(X_train),end='\n\n') print('y_test:\n '+str(X_train),end='\n\n')
- 1 جواب
-
- 1
-

-
ماتزال هذه النسخة تجريبة حتى الآن "experimental" لذا لاستخدامه ، تحتاج إلى استيراد enable_iterative_imputer بشكل صريح لكي يتم استخدام الميزات التجريبية كالتالي: from sklearn.experimental import enable_iterative_imputer from sklearn.impute import IterativeImputer
- 1 جواب
-
- 1
-

-
هي أحد طرق تقسيم الداتا هي متل KFold ولكن يختلف عنه بأنه يقوم بأخذ مجموعات متساوية من حيث عدد الفئات في كل مجموعة في حال كانت البيانات بغرض التنصيف يتم استخدامه عبر الموديل sklearn.model_selection. #استدعاء المكتبات from sklearn.model_selection import StratifiedKFold قمنا باستدعاء المكتبة numpy لتشكيل الداتا. واستدعاء الوظيفه StratifiedKFold من الوحدة model_selection في مكتبة sklearn. #تشكيل الداتا الدخل والخرج X = np.array([[1, 2], [3, 4], [5, 6], [7, 8]]) y = np.array([1, 0, 1, 0]) حيث X مصفوفة أبعادها 4 أسطر وعمودين وy عمود واحد بأربع أسطر. #StratifiedKFold Skf = StratifiedKFold(n_splits=5, *, shuffle=False, random_state=None) الوسيط الأول n_splits عدد صحيح لتحديد عدد الأجزاء folds وهي افتراضية في sklearn من 3 إلى 5. الوسيط الثاني shuffle تأخذ قيمه بوليانية عند وضعها True تقوم بعمل خلط عشوائي للبيانات وfalse عكس ذلك. الوسيط الثالث random_state للتحكم بآلية التقسيم. نفس الأمر في Kfolds مرور حلقة حيث يتم استخدام اثنين من الاندكسات في الحلقة للمرور على التدريب والاختبار وتحوي skf.split(X) هذه الاندكسات . نقوم بطباعة الاندكس للتدريب والاختبار في كل محاوله وبعدها تخزين التدريب والاختبار. ومن ثم طباعة كل منها. #استدعاء المكتبات from sklearn.model_selection import StratifiedKFold #تشكيل الداتا الدخل والخرج X = np.array([[1, 2], [3, 4], [5, 6], [7, 8]]) y = np.array([1, 0, 1, 0]) #StratifiedKFold skf = StratifiedKFold(n_splits=2, shuffle=False, random_state=None) for train_index, test_index in skf.split(X, y): print("TRAIN:", train_index, "TEST:", test_index) X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index] print('X_train:\n '+str(X_train),end='\n\n') print('X_test:\n '+str(X_train),end='\n\n') print('y_train:\n '+str(X_train),end='\n\n') print('y_test:\n '+str(X_train),end='\n\n')
- 1 جواب
-
- 1
-

-
المشكلة في قيم معامل التنعيم C حيث أن القيم المسموح بها هي القيم الموجبة تماماً أي الأكبر تماماً من الصفر، وأنت تحاول تجريب القيمة C=0 وهذا سينتج عنه خطأ. لحل المشكلة يمكنك إعطاء C قيمة متناهية في الصغر بحيث تكون مهملة (أي وكأنها صفر) : from sklearn.svm import SVR from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split from sklearn.model_selection import GridSearchCV import pandas as pd BostonData = load_boston() X = BostonData.data y = BostonData.target X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0, shuffle =True) SelectedModel = SVR() Selected = {'kernel':('linear', 'rbf'),'C': [0.000001, 0.7, 8]} GridSearchModel = GridSearchCV(SelectedModel,Selected, cv = 2,return_train_score=True) GridSearchModel.fit(X_train, y_train)
- 1 جواب
-
- 1
-

-
هو أداة تستخدم لتنظيف البيانات أو معالجة البيانات قبل عملية التدريب، يستخدم في عملية تحديد وجود قيم مفقودة في البيانات حيث يرد مصفوفة بوليانية كل عمود فيها يمثل عمود في البيانات وتحوي فقط الأعمدة التي تحوي قيم مفقودة أي في حال وجود عمود لا يحوي قيم مفقودة لا يقوم بإعطاء عمود له في المصفوفة أما عند وجود قيمة مفقودة في عمود ما يتم إضافة عمود إلى المصفوفة تكون كل قيمه True و مكان القيمة المفقودة false. يتم استخدامه عبر الموديول: sklearn.impute #استدعاء المكتبات: from sklearn.impute import MissingIndicator في البداية قمنا باستدعاء المكتبة التي يوجد فيها MissingIndicator. #الشكل العام MissingIndicator: MI=MissingIndicator(missing_values=nan, features='missing-only', sparse='auto') الوسيط الأول missing_values القيمة المفقودة أي القيمة التي سوف يتم البحث عنها في البيانات و في كثير من الأمثلة تكون القيمة المفقودة Nan أو 0. الوسيط الثاني features في حال كان هذا الوسيط يساوي missing-only بتالي سوف يتم طباعة المصفوفة البوليانة كما ذكرنا سابقا في تعريف MissingIndicator أما في حال all فسوف يتم طباعتها بالكامل مع كافة الأعمدة التي تحوي قيم مفقودة ولا تحوي قيم مفقودة. الوسيط الثالث sparse يتحكم في طباعة المصفوفة إذا كان auto تكون المصفوفة مثل مصفوفة الدخل واذا كان false سوف تكون المصفوفة كاملة أيضا إذا كان true فسوف تكون المصفوفة بدون الميزات التي لا تحوي قيم مفقودة لذلك يتم وضعه auto لترك MissingIndicator يقرر المناسب. طريقة استخدامه: MI=MissingIndicator(missing_values=nan, features='missing-only', sparse='auto') MI.fit_transform(X) حيث الدالة fit_transform يوجد ضمنها جميع العمليات الداخلية لعملية حساب القيم و تطبيقها على البيانات. لنأخذ مثال يوضح MissingIndicator. #استدعاء المكتبات import numpy as np from sklearn.impute import MissingIndicator #تعين داتا دخل مزيفة X = np.array([[np.nan, 2, 3], [0, 1, np.nan], [8, 3, 0]]) #طباعة القيم الناتجه لمعرفة مكان القيم المفقوده MI=MissingIndicator(missing_values=np.nan, features='missing-only',sparse=False) MI.fit_transform(X) #النتيجة array([[ True, False], [False, True], [False, False]])
- 1 جواب
-
- 1
-

-
هي أداة نستخدمها لتطبيق العديد من المعاملات العليا hyperparameters على نموذجنا، لتجريبها و اختيار الأفضل منها. يتم استخدامها عبر الموديول: model_selection.GridSearchCV خطوات تنفيذها: 1. استيراد المكتبة. from sklearn.model_selection import GridSearchCV 2. استيراد الموديل المطلوب فحصه وإنشاؤه. 3. عمل قاموس بحيث يكون فيه المفتاح هو اسم الـ parameter و القيمة هي القيم المطلوب تجريبها له. 4. تنفيذ GridSearchCV حيث نمرر له الموديل المطلوب تنفيذه و القاموس الذي يحوي مانريد نتجريبه. 5. ثم إظهار النتائج عبر عدد من الـ attributes التي سنوردها في المثال (ستكون كل الأمور واضحة جداً) . حيث يجرب GridSearchCV جميع مجموعات القيم التي تم تمريرها في القاموس ويقيم النموذج لكل مجموعة باستخدام طريقة Cross-Validation. ومن ثم، بعد استخدام هذه الوظيفة ، نحصل على الدقة / الخسارة لكل مجموعة من المعلمات العليا ويمكننا اختيار الأفضل أداءً. الصيغة: sklearn.model_selection.GridSearchCV(estimator, param_grid, scoring=None, n_jobs=None, cv=None, verbose=0, return_train_score=False) 1.estimator: النموذج الذي تريد التحقق من المعلمات العليا الخاصة به. 2.params_grid: كائن القاموس الذي يحتوي على المعلمات العليا التي تريد تجربتها. 3-scoring: مقياس التقييم الذي تريد استخدامه. 4.cv: عدد عمليات التحقق المتبادل التي يجب أن تجربها لكل مجموعة مختارة من المعلمات العليا. 5. overbose: يمكنك ضبطه على 1 للحصول على نسخة مطبوعة مفصلة أثناء ملائمة البيانات لـ GridSearchCV . 6.n_jobs: عدد العمليات التي ترغب في تشغيلها بالتوازي لهذه المهمة إذا كانت -1 ستستخدم جميع المعالجات المتاحة. الوسيط الأخير إذا كان False، فلن تتضمن السمة cv_results_ ال score للتدريب. مثال: from sklearn.svm import SVR from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split from sklearn.model_selection import GridSearchCV import pandas as pd # تحميل البيانات BostonData = load_boston() X = BostonData.data y = BostonData.target # تقسيم البيانات X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=0, shuffle =True) #تطبيق GridSearchCV SelectedModel = SVR() ''' :نحدد الآن في القاموس التالي مايلي 1. المفاتيح التي تمثل المعاملات العليا المطلوب إجراء التجريب عليها 2.القيم المراد اختبارها من أجل كل مفتاح لاحظ أننا حددنا للكيرنل قيمتين ليتم تجريبهم وهم linear و rbf :وحددنا للإبسلون القيم التالية 0.1 0.2 0.3 ''' Selected = {'kernel':('linear', 'rbf'), 'epsilon':[0.1,0.2,0.3]} GridSearchModel = GridSearchCV(SelectedModel,Selected, cv = 2,return_train_score=True) # للبدأ بالتدريب GridSearchModel.fit(X_train, y_train) sorted(GridSearchModel.cv_results_.keys()) GridSearchResults = pd.DataFrame(GridSearchModel.cv_results_)[['mean_test_score', 'std_test_score', 'params' , 'rank_test_score' , 'mean_fit_time']] # عرض النتائج print('All Results :', GridSearchResults ) # عرض أفضل نتيجة print('Best Score is :', GridSearchModel.best_score_) # عرض أفضل المعاملات العليا print('Best Parameters :', GridSearchModel.best_params_) #Estimator عرض أفضل print('Best Estimator :', GridSearchModel.best_estimator_) # الخرج ''' All Results : mean_test_score std_test_score params \ 0 0.744228 0.009539 {'epsilon': 0.1, 'kernel': 'linear'} 1 0.234970 0.049682 {'epsilon': 0.1, 'kernel': 'rbf'} 2 0.743469 0.009218 {'epsilon': 0.2, 'kernel': 'linear'} 3 0.232345 0.049925 {'epsilon': 0.2, 'kernel': 'rbf'} 4 0.744154 0.009648 {'epsilon': 0.3, 'kernel': 'linear'} 5 0.231108 0.053474 {'epsilon': 0.3, 'kernel': 'rbf'} rank_test_score mean_fit_time 0 1 0.956472 1 4 0.001997 2 3 1.818950 3 5 0.002998 4 2 1.063380 5 6 0.002010 Best Score is : 0.7442279764447716 Best Parameters : {'epsilon': 0.1, 'kernel': 'linear'} Best Estimator : SVR(C=1.0, cache_size=200, coef0=0.0, degree=3, epsilon=0.1, gamma='scale', kernel='linear', max_iter=-1, shrinking=True, tol=0.001, verbose=False) ''' مثال آخر: # استيراد المكتبات import sklearn from sklearn.svm import SVC from sklearn.model_selection import GridSearchCV from sklearn.model_selection import train_test_split from sklearn.datasets import load_breast_cancer from sklearn.metrics import classification_report, confusion_matrix from sklearn.datasets import load_breast_cancer # تحميل بياناتك dataset = load_breast_cancer() X=dataset.data Y=dataset.target # تقسيمها X_train, X_test, y_train, y_test = train_test_split(X,Y,test_size = 0.2, random_state = 1) # تحديد مانريد اختباره param_grid = {'C': [0.1, 0.7, 8], 'gamma': [1, 0.1, 0.01, 0.0001], 'gamma':['scale', 'auto'], 'kernel': ['linear','rbf']} #GridSearchCV إنشاء ال grid = GridSearchCV(SVC(), param_grid, refit = True,cv=2) # بدأ التدريب والتجريب grid.fit(X_train, y_train) # عرض أفضل المعاملات print(grid.best_params_) # توقع القيم grid_predictions = grid.predict(X_test) #classification_report عرض ال print(classification_report(y_test, grid_predictions)) ''' {'C': 0.1, 'gamma': 'scale', 'kernel': 'linear'} precision recall f1-score support 0 1.00 0.86 0.92 42 1 0.92 1.00 0.96 72 accuracy 0.95 114 macro avg 0.96 0.93 0.94 114 weighted avg 0.95 0.95 0.95 114 '''
- 1 جواب
-
- 1
-

-
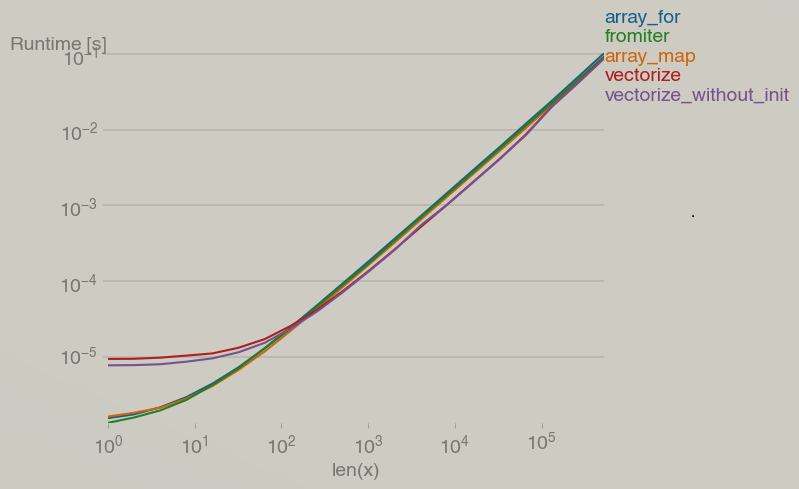
إليك لطرق المستخدمة: import math import numpy x= np.array([0, 1, 2, 3, 4, 5]) g = lambda x: x ** 2 vg = numpy.vectorize(g) def array_for(v): return numpy.array([g(vi) for vi in v]) def array_map(v): return numpy.array(list(map(g, v))) def fromiter(v): return numpy.fromiter((g(vi) for vi in v), v.dtype) def vectorize(v): return numpy.vectorize(g)(v) def vectorize_without_init(v): return vg(v) والاختلاف في التعقيد "complexity" كما في الصورة المرفقة. لاحظ أن التعقيد الزمني يصبح متساوي تقريباً (الفرق في التعقيد يصبح مهمل تقريباً) عندما يزداد طول x عن 2^10.
- 3 اجابة
-
- 2
-

-
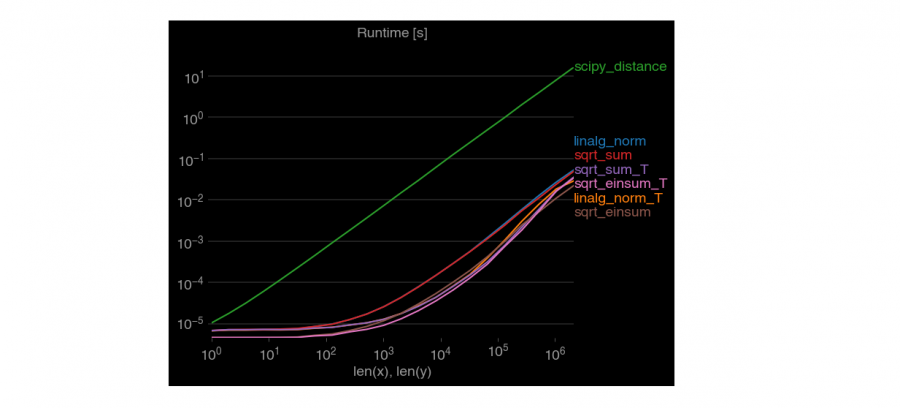
إضافة إلى الطرق التي قدمها الأستاذ وائل. سأضيف لك طريقة هي الأسرع، حيث أنه يوجد العديد من الطرق لحسابها في بايثون و الفرق بينهم هو التعقيد الزمني. حيث يتم استخدام الدالة (Einstein’s summation)einsum كالتالي: import numpy as np point1 = numpy.array((2, 1, 2)) point2 = numpy.array((2, 1, 2)) # التابع التالي سيحسب لك المسافة من أجل نقاط أحادية وثنائية وثلاثية الأبعاد def dist(p1, p2, metric='euclidean'): p1 = np.asarray(p1) p2 = np.atleast_2d(p2) p1_dim = p1.ndim p2_dim = p2.ndim if p1_dim == 1: p1 = p1.reshape(1, 1, p1.shape[0]) if p1_dim >= 2: p1 = p1.reshape(np.prod(p1.shape[:-1]), 1, p1.shape[-1]) if p2_dim > 2: p2 = p2.reshape(np.prod(p2.shape[:-1]), p2.shape[-1]) diff = p1 - p2 dist_arr = np.einsum('ijk,ijk->ij', diff, diff) if metric[:1] == 'e': dist_arr = np.sqrt(dist_arr) dist_arr = np.squeeze(dist_arr) return dist_arr dist(point1,point2) # array(0.) لاحظ التعقيد كيف يختلف من طريقة لأخرى.
- 2 اجابة
-
- 3
-

-
يتم استخدامه عبر الموديول: sklearn.impute #استدعاء المكتبات: from sklearn.impute import KNNImputer في البداية قمنا باستدعاء المكتبة التي يوجد فيها KNNImputer. #الشكل العام KNNImputer: imputerKNN=KNNImputer(missing_values=nan, n_neighbors=5, weights='uniform',metric='nan_euclidean', copy=True) الوسيط الأول missing_values القيمة المفقودة أي القيمه التي سنضع مكانها ما ينوب عنها وفي كثير من الأمثلة تكون القيمه المفقودة Nan أو 0. الوسيط الثاني n_neighbors عدد الجيران الذي سيتم حساب القيمة المتوسطة لهم من أجل عينة تحوي قيمة مفقودة. الوسيط الثالث weights هذا الوسيط يحدد الأساس الذي سيسير عليه KNNImputer حيث يأخذ قيمتين هي ‘uniform’ حيث تعني أن الجار القريب أو البعيد لهم نفس التأثير أي لا يفرق أحدهما في عملية حساب القيمة المتوسطة أما ‘distance’ هنا يتم اعتماد المسافة كعامل تقييم أي الاقرب مسافة إلى المثال الذي يتم التنبؤ بقيمته بتالي سوف يكون تأثير الجار ذو المسافة الأقل أكبر من الجار ذو المسافة الأبعد الوسيط الرابع metric مقياس المسافة للبحث عن الجيران. الوسيط الخامس copy عند وضع هذا الوسيط True يتم أخذ نسخه من البيانات false عكس ذلك أي يتم التطبيق على البيانات الاصلية. مثال: #استدعاء المكتبات import numpy as np from sklearn.impute import KNNImputer #تعين داتا دخل مزيفة X = [[3, 4, np.nan], [3, 4, 3], [np.nan, 1, 2], [8, 8, 7]] #طباعة القيم بعد عملية التنظيف imputerKNN= KNNImputer(n_neighbors=2) imputerKNN.fit_transform(X) #النتيجة array([[3. , 4. , 2.5], [3. , 4. , 3. ], [3. , 1. , 2. ], [8. , 8. , 7. ]]) حيث الدالة fit_transform يوجد ضمنها جميع العمليات الداخلية لعملية حساب القيم و تطبيقها على البيانات.
- 1 جواب
-
- 1
-

-
QDA(Quadratic Discriminant Analysis) هو طريقة تستخدم في تقليل الأبعاد وخاصة في مسائل التصنيف التابعة بالتعليم بإشراف فلو كان لدينا عملية تصنيف ما لأكثر من صنف هو نفس LDA الفرق الوحيد أن LDA عملية الفصل لديه خطية أما QDA مربعة، ويمكن استخدامه كموديل للتنصيف. يتم استخدامه عبر الموديول: sklearn.discriminant_analysis استدعاء المكتبات: from sklearn.discriminant_analysis import QuadraticDiscriminantAnalysis في البداية قمنا باستدعاء المكتبة التي يوجد فيها هذا الموديل. الشكل العام للموديل: QDA=QuadraticDiscriminantAnalysis(priors=None, reg_param=0.0, tol=0.0001) الوسيط الأول priors قيم الاحتمالات للصفوف أي تساوي عدد الصفوف يمكن تمريرها كمصفوفة تحوي الاحتمال لكل صف. الوسيط الثاني reg_param يمثل معامل التنعيم. الوسيط الثالث tol مقدار التسامح في التقارب من القيم الدنيا. طبعا قمت بشرح الشكل العام للموديل ولكن عن طريقة استخدامه سهل جدا فقط بعد أن تقوم بتقسيم الداتا إلى X_train, y_train,X_test, y_test تسطيع كتابة الأتي لعملية التدريب. #الشكل العام للموديل QDA=QuadraticDiscriminantAnalysis( priors=None, reg_param=0.0,tol=0.0001) QDA.fit(X_train, y_train) حيث الدالة fit يوجد ضمنها جميع العمليات الداخلية لعملية التدريب. يوجد دالة أخرى تستخدم لغرض التنبؤ كالاتي: #حساب القيم المتوقعة y_pred =َQDA.predict(X_test) نستطيع حساب دقة الموديل أو كفاءته على التدريب والاختبار عن طريق التابع score ويكون وفق الشكل: # حساب الكفاءه على الاختبار والتدريب print('QDA Train Score is : ' , QDA.score(X_train, y_train)) print('QDA Test Score is : ' , QDA.score(X_test, y_test)) لنأخذ مثال يوضح المصنف: قمنا باستدعاء المكتبات وبناء عينة مزيفه وكان التصنيف ثنائي أما 1 أو 0. ثم قمنا بتجريب الموديل على عينة معطاة. # استدعاء المكتبات import numpy as np from sklearn.discriminant_analysis import QuadraticDiscriminantAnalysis #تعين داتا دخل مزيفة X = np.array([[1, 1], [3, 1], [-3, -2], [-1, -1], [-3, -1], [3, 2]]) y = np.array([1, 0, 0, 1, 1, 1]) #بناء الموديل QDA = QuadraticDiscriminantAnalysis() QDA.fit(X,y) #طباعة تصنيف العينه print(QDA.predict([[-1, -0.4]])) #النتيجة #[1]
- 1 جواب
-
- 1
-

-
LDA(Linear Discriminant Analysis) هي طريقة تستخدم في تقليل الأبعاد وخاصة في مسائل التصنيف التابعة بالتعليم بإشراف فلو كان لدينا عملية تصنيف ما لأكثر من صنف وكان تمثيل الداتا بالمستوى ثنائي الابعاد فالخط المستقيم يمكن أن يكون غير كافي في عملية الفصل لذلك يتم استخدام LDA ليقوم بتحويل المستوي ثنائي الابعاد إلى أحادي الأبعاد. ويمكن استخدامه كموديل للتنصيف. يتم استخداه عبر الموديول: sklearn.discriminant_analysis استدعاء المكتبات: from sklearn.discriminant_analysis import LinearDiscriminantAnalysis في البداية قمنا باستدعاء المكتبة التي يوجد فيها هذا الموديل. # الصيغة المبسطة للموديل: LDA=LinearDiscriminantAnalysis(solver='svd', shrinkage=None, priors=None, n_components=None, tol=0.0001) الوسيط الأول solver يمثل طريقة الحل ويأخذ ثلاث قيم {‘svd’, ‘lsqr’, ‘eigen’} الوسيط الثاني shrinkage معامل تنعيم يستخدم لتحسين تقدير المصفوفات في حال كان عدد عينات التدريب صغير وعدد الميزات كبير الوسيط الثالث priors قيم الاحتمالات للصفوف أي تساوي عدد الصفوف يمكن تمريرها كمصفوفة تحوي الاحتمال لكل صف الوسيط الرابع n_components هو عدد حقيقي أو صحيح يشير إلى عدد المكونات التي سيتم الإبقاء عليها المقصود بالمكونات أٌقل عدد أمثلة وعدد الفيتشرز وإذا أخذ None يتم الاحتفاظ بجميع المكونات الوسيط الخامس tol مقدار التسامح في التقارب من القيم الدنيا طبعا قمت بشرح الشكل العام للموديل ولكن عن طريقة استخدامه سهل جداً فقط بعد أن تقوم بتقسيم الداتا إلى X_train, y_train,X_test, y_test تسطيع كتابة الأتي لعملية التدريب # الشكل العام للموديل: LDA=LinearDiscriminantAnalysis(solver='svd', shrinkage=None, priors=None, n_components=None, tol=0.0001 ) LDA.fit(X_train, y_train) حيث الدالة fit يوجد ضمنها جميع العمليات الداخلية لعملية التدريب. يوجد دالة أخرى تستخدم لغرض التنبؤ كالاتي.. #حساب القيم المتوقعة: y_pred =LDA.predict(X_test) نستطيع حساب دقة الموديل أو كفاءته على التدريب والاختبار عن طريق التابع score ويكون وفق الشكل: حساب الكفاءة على الاختبار والتدريب: print('LDA Train Score is : ' , LDA.score(X_train, y_train)) print('LDA Test Score is : ' , LDA.score(X_test, y_test)) لنأخذ مثال يوضح المصنف.. قمنا باستدعاء المكتبات وبناء عينة مزيفة وكان التصنيف ثنائي أما 1 أو 0. ثم قمنا بتجريب الموديل على عينة معطاة. استدعاء المكتبات import numpy as np from sklearn.discriminant_analysis import LinearDiscriminantAnalysis #تعين داتا دخل مزيفة X = np.array([[1, 1], [3, 1], [-3, -2], [-1, -1], [-3, -1], [3, 2]]) y = np.array([0, 0, 1, 1, 0, 1]) #بناء الموديل LDA = LinearDiscriminantAnalysis() LDA.fit(X,y) #طباعة تصنيف العينه print(LDA.predict([[-1, -0.4]])) #النتيجة #[0]
- 2 اجابة
-
- 1
-

-
الخطأ الذي ظهر لك هو نتيجة غير مباشرة لاستخدامك نموذج توقع ك base_estimator في نموذج BaggingClassifier، حيث أن المهمة التي تعمل عليها هي مهمة تصنيف والنموذج BaggingClassifier هو نموذج تصنيف وتحاول استخدام نموذج SVR وهو نموذج توقع ك base_estimator وهذا خاطئ، يجب استخدام نموذج تصنيف حصراً ك base_estimator. لإصلاح الأمر استخدم نموذج تصنيف مثل SVC أو قم بضبط ال base_estimator على None.
- 2 اجابة
-
- 1
-

-
سأعرف لك تابع عام يقوم باستبدال نص بنص آخر، ٍسأتخدم االمكتبة regex هنا: import re search_text="2 hi there" new_text="hi" def replaceText(path, oldtext, newtext, flags=0 ): with open( path, "r+" ) as f: content = f.read() pa = re.compile( re.escape( oldtext ), flags ) content = pa.sub( newtext, content ) f.seek( 0 ) f.truncate() f.write( content ) # قبل الاستبدال path="D:\\ff.txt" f = open(path, "r") print(f.read()) ''' 1 hi there 2 hi there 3 hi there ''' replaceText(path,search_text,new_text) # بعد الاستبدال f = open(path, "r") print(f.read()) ''' 1 hi there hi 3 hi there ''' يمكنك أيضاً استخدام الوحدة fileinput التي تحوي على توابع تسمح بتسريع التعامل مع هذه الأمور: import fileinput search_text="2 hi there" new_text="hi" def replaceText(path, oldtext, newtext): with fileinput.input(path, inplace=1) as file: for line in file: new = line.replace(oldtext, newtext) print(new, end='') # قبل الاستبدال path="D:\\ff.txt" f = open(path, "r") print(f.read()) ''' 1 hi there 2 hi there 3 hi there ''' f.close() replace_in_file(path,search_text,new_text) # بعد الاستبدال f = open(path, "r") print(f.read()) ''' 1 hi there hi 3 hi there '''
- 4 اجابة
-
- 2
-

-
السبب يعود إلى مبدأ "Command–query separation" حيث يعد الفصل بين الأوامر والاستعلام مبدأ في برمجة الكمبيوتر. ينص هذا المبدأ على التالي:""إن كل عملية يجب أن تكون إما أمرًا يقوم بتنفيذ إجراء ما ، أو استعلام يقوم بإرجاع البيانات ، وليس كليهما. وفي بايثون يتم تحقيق هذا المبدأ في التابع append والعديد من التوابع الإجرائية الأخرى (ليست استعلام). ولهذا السبب أعادت لك None أي لاشيء. وبالتالي not None تكافئ True. لكن قد يكون هناك بعض التجاوزات لهذا المبدأ مثل التابع pop الذي يعد تابع إجرائي وبنفس الوقت يعيد قيمة، حيث يحذف آخر عنصر من list ويرجعه بنفس الوقت: list = [ 1, 2, 3, 4, 5, 6 ] print(list.pop()) # 6 print(list)# [1, 2, 3, 4, 5]
- 4 اجابة
-
- 1
-

-
أنا استخدم مكتبة dpath وهي سريعة بما يكفي ومصممة خصيصاً للتعامل مع هكذا مهام في القواميس كالتالي: # أولاً قم بتحميلها # pip install dpath dataDict = { "a":{ "x": 1, "y": 2, "z": 3 }, "b":{ "x": 1, "y": { "x": 1, "y": 2, "z": 3 }, "z": 3 } } # استيراد المكتبة import dpath.util # البحث عن القيمة المطلوبة value1=dpath.util.get(dataDict, '/a/z') # نمرر القاموس والمسار المطلوب print(value1) value1=dpath.util.get(dataDict, '/b/y/z') print(value2) هناك طرق أخرى، مثلاً يمكنك استخدام الدالة reduce من الموديول functools كالتالي: from functools import reduce import operator def getFromDict(dataDict, mapList): return reduce(operator.getitem, mapList, dataDict) dataDict = { "a":{ "x": 1, "y": 2, "z": 3 }, "b":{ "x": 1, "y": { "x": 1, "y": 2, "z": 3 }, "z": 3 } } value1=getFromDict(dataDict, ["a","z"]) print(value1) value2=getFromDict(dataDict, ["b","y","z"]) print(value2)
- 4 اجابة
-
- 1
-

-
لانستخدم الترميز One-Hot مع توابع حساب كفاءة النموذج في Sklearn. المشكلة في أن مصفوفة الCM لاتقبل أن تكون مدخلاتها من الشكل One-Hot، وإنما يجب أن تكون كالتالي: y_true (n_samples,) y_predict(n_samples,) وبالتالي لحل مشكلتك يجب أن لاتستخدم التحويل One-Hot مع المصفوفة CM. إذا أردت إرجاع بياناتك لشكلها الطبيعي (بدون ال One-Hot) يمكنك استخدام التابع argmax على للبيانات التي حولتها إلى الترميز One-Hot كالتالي: y=y.argmax(axis=1) ثم يمكنك حساب ال CM بدون مشاكل. إليك مثال: # هكذا تتوقع المصفوفة أن تكون مدخلاتها from sklearn.metrics import confusion_matrix y_true = [1, 1, 0, 2, 0, 1] y_pred = [1, 0, 2, 2, 0, 2] confusion_matrix(y_true, y_pred)
- 2 اجابة
-
- 1
-

-
بما أنك تستخدم الوسيط stratify (الطبقات)، فهذا يتطلب أن يكون عدد العينات التي تنتمي إلى كل فئة متناسب في كل من بيانات التدريب والاختبار. لكن لديك فئة من بياناتك تحتوي على عينة واحدة فقط. لذلك إما أن تكون في بيانات التدريب أو الاختبار في وقت واحد وهذا يخالف خيار التقسيم الطبقي. لذالك ينتج الخطأ. يمكنك حل المشكلة في أن تزيد عدد البيانات التي تنتمي لهذا الصنف، أو أن تلغي خاصية التقسيم الطبقي.
- 2 اجابة
-
- 1
-

-
قد يظهر هذا الخطأ في أي نموذج آخر في Sklearn بشكل مشابه تماماً. المشكلة تظهر في التابع fit ، حيث أن التابع يتوقع أبعاد X بالشكل: [n_samples,n_features] لكنك تعطيه مصفوفة من الشكل: [n_samples,] لحل المشكلة قم بإعادة تعيين الأبعاد: X=X.reshape(-1,1) lr.fit(X ,y)
- 2 اجابة
-
- 1
-

-
أنت لديك 5 عينات، موزعة كالتالي: عينة للأصناف 2و1و3 وعينتين للصنف 4 ال "number of members in each class" يقصد بها عدد الأعضاء في كل صف أي عدد العينات من أجل كل صف. يحاول StratifiedKFold الحفاظ على نسبة معينة في كل fold من هؤلاء الأعضاء. أنت حددت 3 تقسيمات "Fold" وبالتالي في كل تقسيمة من أجل الكلاس 2 مثلاً، يجب على الأقل الحفاظ على نسبة0.33 =1/3 عضو. ومن الكلاس 1 و 3 أيضاً يجب أن تتحقق نفس النسبة (0.33 عضو). أما من أجل الكلاس 4 فيجب أن تتحقق النسبة 2/3=0.67 عضو. لكن 0.33 تعني جزء من العينة وهذا غير ممكن! يجب على الأقل أن يكون عدد الأعضاء في الكلاسات من 1 إلى 3 يساوي 3 ومن الكلاس 4 أيضاً 3 لكي يصبح 1 عضو في كل تقسيمة على الأقل. ولهذا السبب ظهر الخطأ. إذاً يجب أن يكون لديك على الأقل 3 عينات من أجل كل كلاس. لاحظ أيضاً أنك إذا وضعت 2 (أقصد تقسيمتين) سوف ينجح الأمر لكنه سيعطيك التحذير التالي: UserWarning: The least populated class in y has only 1 members, which is less than n_splits=2. % (min_groups, self.n_splits)), UserWarning) حيث أنه يعطيك خطأ إذا لم يجد أي كلاس يحقق الشرط (لأنه لن يكون لفكرة الخوارزمية معنى بعد ذلك)، بينما يعطيك تحذير إذا كان هناك كلاس واحد على الأقل يحققه.
- 2 اجابة
-
- 2
-

-
تقوم على تحديد عدد معين من التقسيمات وفي كل تقسيمة يتم اختيار نسبة معينة من البيانات عشوائياً للاستخدام كعينة اختبار والباقي للتدريب. سأقوم بتطبيقه على نموذج تصنيف لكي تعرف كيفية تطبيقه بسكل عملي: from sklearn.model_selection import ShuffleSplit from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score from sklearn.datasets import make_blobs # إنشاء داتاسيت مزيفة ب10 عينات و3 فئات X, y = make_blobs(n_samples=10, random_state=2021) # y=array([1, 1, 2, 0, 1, 2, 0, 2, 0, 0]) # ShuffleSplit إنشاء كائن من الكلاس # حددنا عدد التقسيمات ب 5 وهو العدد الافتراضي # وحددنا حجم عينة الاحتبار ب20 في المئة cv = ShuffleSplit(n_splits=5, test_size=0.2, random_state=0) # لتخزين النتائج من كل تقسيمة y_true, y_pred = list(), list() # عدد التقسيمات sp=cv.get_n_splits(X) # عملية التقسيم for train_ix, test_ix in cv.split(X): # تقسيم البيانات X_train, X_test = X[train_ix, :], X[test_ix, :] y_train, y_test = y[train_ix], y[test_ix] # تدريب النموذج model = RandomForestClassifier(random_state=44) model.fit(X_train, y_train) # توقع النموذج yhat = model.predict(X_test) # تخزين النتيجة for i in range(0,len(y_test)): y_true.append(y_test[i]) y_pred.append(yhat[i]) # حساب الدقة acc = accuracy_score(y_true, y_pred) print('Accuracy: %.3f' % acc)# Accuracy: 1.000 لاحظ كيف أنه قمنا بتحديد عدد معين من التقسيمات وهو 5 وحددنا نسبة مئوية للعينات التي سيتم استخدامها للاختبار في كل تقسيمة وهو 20%، ثم أدرب النموذج على بيانات التدريب وأقوم بحساب القيمة المتوقعة على عينات الاختبار وأضعها في y_pred وأضع القيم الحقيقية المقابلة لها في y_true, وهكذا بالنسبة لبقية التقسيمات حتى ننتهي وبعدها أقوم بحساب دقة النموذج.
- 2 اجابة
-
- 1
-

-
بشكل مشابه ل LeaveOneOut لكن مع اختلاف بسيط. هذه الطريقة تقوم على تقييم النموذج على كامل العينات، بحيث في كل مرة تقوم بتدريب النموذج على كل العينات ماعدا عدد محدد من العينات تخرجها لكي تقوم باستخدامها للاختبار. المثاليين يوضحان كل شيء: قم بتشغيل الكود لترى الخرج مباشرة. import numpy as np from sklearn.model_selection import LeavePOut X = np.array([[1,4],[2,1],[3,4],[7,8]]) y = np.array([2,1,3,9]) #3 هنا حددنا عدد العينات lpo = LeavePOut(3) # لمعرفة عدد التقسيمات الممكنة print(lpo.get_n_splits(X)) # تقسيم البيانات for train_index, test_index in lpo.split(X): # للتقسيمة index عرض ال print("TRAIN:"+str(train_index)+'\n'+"TEST:"+str(test_index),end='\n\n') # تقسيم البيانات X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index] # عرض البيانات المقسمة print('X_train:\n '+str(X_train),end='\n\n') print('X_test:\n '+str(X_test),end='\n\n') print('y_train:\n '+str(y_train),end='\n\n') print('y_test:\n' +str(y_test),end='\n\n') في المثال التالي سوف استخدم هذا النهج في تدريب نموذج واختباره: from sklearn.model_selection import LeavePOut from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score from sklearn.datasets import make_blobs # إنشاء داتاسيت مزيفة ب10 عينات و3 فئات X, y = make_blobs(n_samples=10, random_state=2021) # y=array([1, 1, 2, 0, 1, 2, 0, 2, 0, 0]) # LeavePOut إنشاء كائن من الكلاس # تحديد عدد العينات التي نريدها للاختبار في كل مرة pout=2 cv = LeavePOut(p=pout) # لتخزين النتائج من كل تقسيمة y_true, y_pred = list(), list() # عملية التقسيم for train_ix, test_ix in cv.split(X): # تقسيم البيانات X_train, X_test = X[train_ix, :], X[test_ix, :] y_train, y_test = y[train_ix], y[test_ix] # تدريب النموذج model = RandomForestClassifier(random_state=44) model.fit(X_train, y_train) # تقييم النموذج yhat = model.predict(X_test) # تخزين النتيجة for i in range(0,pout): y_true.append(y_test[i]) y_pred.append(yhat[i]) # حساب الدقة acc = accuracy_score(y_true, y_pred) print('Accuracy: %.3f' % acc)# Accuracy: 1.000 لاحظ كيف أنه في كل مرة أقوم بأخذ عدة عينات (حددها بشكل اختياري) للاختبار والباقي للتدريب ثم أدرب النموذج عليها وأقوم بحساب القيمة المتوقعة على عينات الاختبار وأضعها في y_pred وأضع القيم الحقيقية المقابلة لها في y_true, وهكذا بالنسبة لبقية التقسيمات حتى ننتهي وبعدها أقوم بحساب دقة النموذج.
- 3 اجابة
-
- 1
-

-
تم تقديم عبارة with لأول مرة منذ خمس سنوات، في Python 2.5. تُستخدم with عند العمل مع موارد غير مُدارة "unmanaged resources" مثل file streams Open network connections. Unmanaged memory ومع الأقفال Locks و ال sockets وال subprocesses. يسمح لك بالتأكد من "تنظيف" المورد عند انتهاء تشغيل الكود الذي يستخدمه، حتى إذا تم طرح استثناءات. ففي حال استخدمتها مع الملفات فتتمثل ميزة استخدام عبارة with في ضمان إغلاق الملف بشكل آمن بغض النظر عن كيفية الخروج من الكتل البرمجية المتداخلة الموجودة لديك. بحيث إذا حدث استثناء قبل نهاية الكتلة البرمجية، فسيتم إغلاق الملف بشكل مسبق بواسطة معالج استثناء خارجي. وإذا كانت الكتلة المتداخلة تحتوي على تعليمة return ، أو تعليمة continue أو break، فإن تعليمة with ستغلق الملف تلقائياً في تلك الحالات أيضاً. حيث تضمن عبارة with نفسها بالحصول على الموارد وتحريرها بالشكل المناسب. يكون استخدامها مفيداً عندما يكون لديك عمليتان مترابطتان ترغب في تنفيذهما كزوج، مع وجود كتلة من التعليمات البرمجية بينهما. المثال الكلاسيكي هو فتح ملف ومعالجة الملف ثم إغلاقه وهذا ماسنراه في المثال التالي: # وبدون معالجة للاستثناءات التي قد تحدث with بدون استخدام f = open('path', 'w') f.write('hsoub') f.close() # مع معالجة للاستثناءات التي قد تحدث with بدون استخدام f = open('path', 'w') try: f.write('hsoub') finally: f.close() #with استخدام with open('path', 'w') as file: f.write('hsoub') في أول مثال قد يؤدي حدوث استثناء أثناء استدعاء write إلى عدم إغلاق الملف بشكل سليم مما يؤدي إلى حدوث العديد من الأخطاء في الكود. الطريقة الثانية في المثال أعلاه تهتم بجميع الاستثناءات ولكن استخدام تعليمة with يجعل الكود مضغوطاً وقابل للقراءة بشكل أكبر. وبالتالي ، تساعد العبارة في تجنب الأخطاء والتسريبات من خلال ضمان تحرير المورد بشكل صحيح عند تنفيذ التعليمات البرمجية التي تستخدم المورد بالكامل. ولاحظ أنك لن تحتاج لتعليمة close كما في أول حالتين. يمكنك أيضاً استخدام تعليمة with مع كائنات معرفة من قبلك حيث يمكن استخدامها في الكائنات التي يحددها المستخدم وهذا مفيد بالنسبة لك لأن دعم عبارة with في العناصر الخاصة بك سيضمن عدم ترك أي مورد مفتوحًا أبدًا. لاستخدامها مع الكائنات المعرفة من قبل المستخدم، تحتاج فقط إلى إضافة التوابع __enter __ () و __exit __ () في الكائن، مثال: class wr(object): def __init__(self, file_name): self.file_name = file_name def __enter__(self): self.file = open(self.file_name, 'w') return self.file def __exit__(self): self.file.close() #مع الكائن with استخدام التعليمة with wr('file.txt') as f: f.write('hasoub') إن الكلمة المفتاحية with تشكل باني ل wr، وبمجرد وصول التنفيذ لتعليمة with يتم إنشاء كائن من wr، ثم يقوم بايثون باستدعاء التابع enter الذي يقوم بتهيئة المورد الذي تريد أن تستخدمه في ال object الخاص بك، ويجب أن تقوم طريقة __enter __ () دائمًا بإرجاع واصف للمورد "descriptor"(مقبض للوصول للملف) الذي تم الحصول عليه. يتم استخدام f للإشارة لل descriptor الذي تم الحصول عليه من التابع enter، ويتم وضع الكود البرمجي الذي يستخدم المورد بداخل كتلة with وبمجرد تنفيذ الكود الموجود داخل الكتلة with ، يتم استدعاء طريقة __exit __ () ليتم تحرير جميع الموارد. # وهذا ليس كل شيء عن تعليمة with.
- 4 اجابة
-
- 2
-

-
الكلاس Imputer تم حذفه من الموديول preprocessing في النسخ الحديثة من Sklearn وتم تضمينه في الموديول impute، لذا يجب أن تقوم باستيراده بالشكل التالي : from sklearn.impute import SimpleImputer ويمكنك أيضاً استيراد الأنواع الأخرى بنفس الطريقة: from sklearn.impute import KNNImputer from sklearn.impute import MissingIndicator from sklearn.impute import IterativeImputer
- 2 اجابة
-
- 2
-

-
هذه المشكلة قد تظهر معك في حالة استخدمت أي خوارزمية أخرى في Sklearn. المشكلة تظهر عندما يصل التنفيذ للتابع fit. السبب في أن بيانات y_train من النمط object لذلك فإن Sklearn لم تستطع التعرف على نوعها. ولحل المشكلة قم بتحويل بيانات ال y_train إلى النمط integer كالتالي: y_train =y_train .astype('int')
- 1 جواب
-
- 1
-