-
المساهمات
1068 -
تاريخ الانضمام
-
تاريخ آخر زيارة
-
عدد الأيام التي تصدر بها
43
نوع المحتوى
ريادة الأعمال
البرمجة
التصميم
DevOps
التسويق والمبيعات
العمل الحر
البرامج والتطبيقات
آخر التحديثات
قصص نجاح
أسئلة وأجوبة
كتب
دورات
كل منشورات العضو Ali Haidar Ahmad
-
هذه الطريقة تقوم على تقييم النموذج على كامل العينات، بحيث في كل مرة تقوم بتدريب النموذج على كل العينات ماعدا عينة واحدة تخرجها لكي تقوم باستخدامها للاختبار. المثاليين يوضحان كل شيء: import numpy as np from sklearn.model_selection import LeaveOneOut X = np.array([[1,4],[2,1],[3,4],[7,8]]) y = np.array([2,1,3,9]) loo = LeaveOneOut() # لمعرفة عدد التقسيمات الممكنة print(loo.get_n_splits(X)) # تقسيم البيانات for train_index, test_index in loo.split(X): # للتقسيمة index عرض ال print("TRAIN:"+str(train_index)+'\n'+"TEST:"+str(test_index),end='\n\n') # تقسيم البيانات X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index] # عرض البيانات المقسمة print('X_train:\n '+str(X_train),end='\n\n') print('X_test:\n '+str(X_test),end='\n\n') print('y_train:\n '+str(y_train),end='\n\n') print('y_test:\n' +str(y_test),end='\n\n') #--------------------------------------------------------------------------------------------------# 4 TRAIN:[1 2 3] TEST:[0] X_train: [[2 1] [3 4] [7 8]] X_test: [[1 4]] y_train: [1 3 9] y_test: [2] TRAIN:[0 2 3] TEST:[1] X_train: [[1 4] [3 4] [7 8]] X_test: [[2 1]] y_train: [2 3 9] y_test: [1] TRAIN:[0 1 3] TEST:[2] X_train: [[1 4] [2 1] [7 8]] X_test: [[3 4]] y_train: [2 1 9] y_test: [3] TRAIN:[0 1 2] TEST:[3] X_train: [[1 4] [2 1] [3 4]] X_test: [[7 8]] y_train: [2 1 3] y_test: [9] لاحظ كيف أنه في كل تكرار يتم أخذ عينة واحدة للاختبار وباقي العينات للتدريب. في المثال التالي سوف أستخدم هذا النهج في تدريب نموذج واختباره: from sklearn.model_selection import LeaveOneOut from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score from sklearn.datasets import make_blobs # إنشاء داتاسيت مزيفة ب10 عينات و3 فئات X, y = make_blobs(n_samples=10, random_state=2021) # y=array([1, 1, 2, 0, 1, 2, 0, 2, 0, 0]) # LeaveOneOut إنشاء كائن من الكلاس cv = LeaveOneOut() # لتخزين النتائج من كل تقسيمة y_true, y_pred = list(), list() # عملية التقسيم for train_ix, test_ix in cv.split(X): print("TRAIN:"+str(train_ix)+'\n'+"TEST:"+str(test_ix),end='\n\n') # تقسيم البيانات X_train, X_test = X[train_ix, :], X[test_ix, :] y_train, y_test = y[train_ix], y[test_ix] # تدريب النموذج model = RandomForestClassifier(random_state=44) model.fit(X_train, y_train) # تقييم النموذج yhat = model.predict(X_test) # تخزين النتيجة y_true.append(y_test[0]) y_pred.append(yhat[0]) # حساب الدقة acc = accuracy_score(y_true, y_pred) print('Accuracy: %.3f' % acc) لاحظ كيف أنه في كل مرة أقوم بأخذ عينة واحدة للاختبار والباقي للتدريب ثم أدرب النموذج عليها وأقوم بحساب القيمة المتوقعة على عينة الاختبار وأضعها في y_pred وأضع القيمة الحقيقية في y_true, وهكذا بالنسبة لبقية التقسيمات حتى ننتهي وبعدها أقوم بحساب دقة النموذج.
- 1 جواب
-
- 1
-

-
هذا المنهج هو نهج من مناهج ال Feature Selection ويعمل على حذف ال Feature التي يكون تباينها أقل من عتبة محددة.حيث يزيل بشكل افتراضي كل ال Features التي يكون تباينها صفري (نفس القيم في كل العينات). المثال التالي سيجعل الأمر واضح: قمت بإنشاء مجموعة بيانات صغيرة وقمت بجعل ال feature الأولى منها ذات تباين قليل (أغلب قيمها أصفار)، ثم قمت بتطبيق عتبة threshold وقمت بتمريرها للكلاس VarianceThreshold بحيث أن شكل العتبة سأجعله من الشكل: p(1-p) في حالة وضعنا p=8 كما في مثالنا، فسيتم حذف ال Features التي تحوي قيم مكررة بنسبة أكبر أو تساوي 80%. p=10 فسيتم حذف ال Features التي تحوي قيم مكررة بنسبة تساوي 100%. from sklearn.feature_selection import VarianceThreshold X = [[0, 0, 1], [0, 1, 0], [1, 0, 0], [0, 1, 1], [0, 1, 0], [0, 1, 1]] print(X) # هنا سنحذف الفيتشرز التي تتكرر قيمها بنسبة أكثر من ثمانون بالمئة sel = VarianceThreshold(threshold=(.8 * (1 - .8))) X=sel.fit_transform(X) print(X) ''' [[0 1] [1 0] [0 0] [1 1] [1 0] [1 1]] ''' لاحظ كيف تم حذف أول feature. مثال آخر: from sklearn.feature_selection import VarianceThreshold X = [[0, 0, 1,3], [0, 1, 0,3], [1, 0, 0,3], [0, 1, 1,3], [0, 1, 0,3], [0, 1, 1,3]] print(X) # هنا سنحذف الفيتشرز التي تتكرر قيمها بنسبة مئة بالمئة sel = VarianceThreshold(threshold=(.10 * (1 - .10))) X=sel.fit_transform(X) print(X) ''' [[0 0 1] [0 1 0] [1 0 0] [0 1 1] [0 1 0] [0 1 1]] '''
- 1 جواب
-
- 1
-

-
يمكنك استخدام الكلاس QuantileTransformer من الموديول: sklearn.preprocessing.QuantileTransformer في المثال التالي سأوضح لك الأمر: قمت بإنشاء مجموعة بيانات صغيرة موزعة توزيعاً غاوصياً، ثم قمت بجعل قيم هذه البيانات متطرفة عن طريق إدخالها بتابع أسي، ثم بعد ذلك سنطبق التحويل QuantileTransformer لكي نعيد نوحيد البيانات وجعلها موزعة توزيعاً موحداً uniform. from numpy.random import randn from sklearn.preprocessing import QuantileTransformer from numpy import exp from matplotlib import pyplot # توليد بيانات غاوصية data = randn(2000) # نغير القيم في بياناتنا بحيث نجعل قيمها متطرفة data = exp(data) # نرسك شكل البيانات بعد جعلها متطرفة pyplot.hist(data, bins=50) pyplot.show() # عمل إعادة تعيين لأبعاد البيانات للحصول على أسطر وأعمدة data = data.reshape((len(data),1)) #quantile transform تطبيق تحويل quantile = QuantileTransformer(n_quantiles=2000,output_distribution='uniform',copy=True) data = quantile.fit_transform(data) # عرض شكل البيانات بعد تطبيق التحويل pyplot.hist(data, bins=50) pyplot.show() الوسيط الأول: عدد الكميات المطلوب حسابها. إنه يتوافق مع عدد المعالم المستخدمة لتحديد دالة التوزيع التراكمي. إذا كانت n_quantiles أكبر من عدد العينات ، فسيتم تعيين n_quantiles على عدد العينات نظرًا لأن عددًا أكبر من الكميات لا يعطي تقديرًا تقريبيًا أفضل لمقدر دالة التوزيع التراكمي. افتراضياً 1000. الوسيط الثاني: لتحديد نظام توحيد البيانات المطلوب (إما توزيع طبيعي-غاوصي- أو توزيع موحد كما في مثالنا) أي إما normal أو uniform. الثالث: لكل لايتم تطبيق التغييرات على البيانات الأساسية.
- 1 جواب
-
- 1
-

-
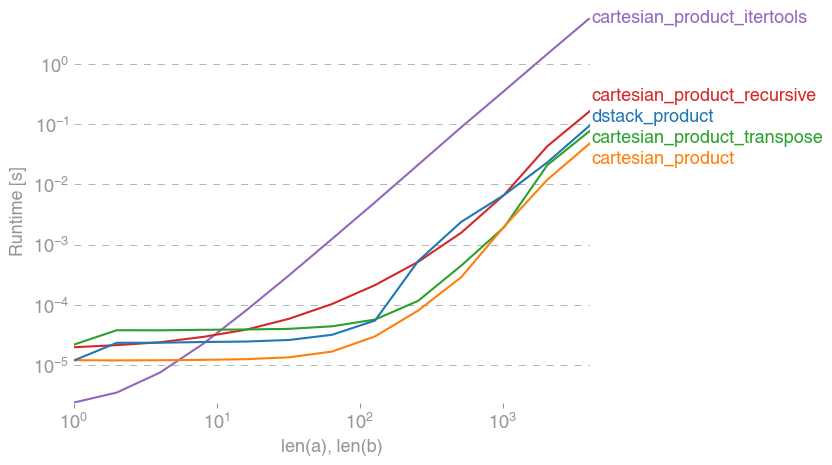
إليك طريقة أخرى تعتبر الأسرع : a = numpy.array([0,1, 2]) b = numpy.array([3, 4, 5]) def cp(*arrays): le = len(arrays) arr = np.empty([len(a) for a in arrays] + [le], dtype=np.result_type(*arrays)) for j, x in enumerate(np.ix_(*arrays)): arr[...,j] = x return arr.reshape(-1, le) cp(a,b) ''' array([[0, 3], [0, 4], [0, 5], [1, 3], [1, 4], [1, 5], [2, 3], [2, 4], [2, 5]]) ''' لاحظ في الصورة أدناه مقارنة بين عدة طرق لتحقيق الجداء الديكارتي (المنحنى ذو اللون البرتقالي يمثل تابعنا والأخضر يمثل استخدام ال transpose) لاحظ أيضاً أن أسوأ طريقة هي كما أشرت استخدام "itertools"
- 4 اجابة
-
- 1
-

-
import pandas as pd df = pd.DataFrame({ 'Type': ['A', 'B', 'O', 'B'], 'Set': ['A', 'B', 'B', 'A'] }) # طباعة الداتا الأساسية قبل التعديل print("Initial DataFrame:") print(df, "\n") #apply إضافة العمود الجديد اعتماداً على تطبيق التابع df['newcol'] = df.apply(lambda df:"group1" if (df.Set=="A") else "group2",axis=1) # طباعة الشكل الجديد للداتا print("DataFrame after addition of new column") print(df, "\n") ''' ______________________________________________________________________________________ Initial DataFrame: Type Set 0 A A 1 B B 2 O B 3 B A DataFrame after addition of new column Type Set newcol 0 A A group1 1 B B group2 2 O B group2 3 B A group1 ''' يمكنك القيام بذلك كما ترى في المثال عن طريق استخدام التابع apply من مكتبة pandas مع التعبير lambda حيث نجعل متحولاً (df في مثالنا) يمر على كل سطر في ال dataframe ويختبر القيمة الموجودة في العمود المطلوب ونجعله يقرر القيمة التي سنضعها في العمود الجديد على أساس هذا الشرط، ولاتنسى أن تجعل ال axis =1 لكي يتم تطبيق التابع على كل سطر
- 4 اجابة
-
- 1
-

-
نستخدم هذه الأداة لقياس ال score لنموذج أو عدة نماذج في كل Folds. يمكنك استخدامها عبر الموديول: sklearn.model_selection.cross_val_score الصيغة المبسطة: sklearn.model_selection.cross_val_score(estimator, X, y, cv=None) الوسيط cv لتحديد عدد ال Folds التي نريد تطبيقها. ال estimator هو الخوارزمية المطلوب تطبيقها. مثال للتوضيح (استخدام مودل واحد): from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split from sklearn.model_selection import cross_val_score from sklearn.ensemble import RandomForestRegressor # تحميل الداتا BostonData = load_boston() data = BostonData.data labels = BostonData.target # تقسيم البيانات X_train, X_test, y_train, y_test = train_test_split(data, labels, test_size=0.2, shuffle =True, random_state=2021) CVTrain = cross_val_score(RandomForestRegressor(), X_train, y_train, cv=5) CVTest = cross_val_score(RandomForestRegressor(), X_test, y_test, cv=5) # عرض النتائج print('Cross Validate Score for Training Set: ', CVTrain) print('Cross Validate Score for Testing Set: ', CVTest) # Cross Validate Score for Training Set: [0.88749657 0.885243 0.90868134 0.89021845 0.81435844] # Cross Validate Score for Testing Set: [0.68090613 0.84052288 0.7597606 0.49063984 0.66992151] استخدام عدة نماذج: from sklearn.datasets import load_boston from sklearn.tree import DecisionTreeRegressor from sklearn.ensemble import RandomForestRegressor from sklearn.svm import SVR from sklearn.model_selection import cross_val_score # تحميل الداتا BostonData = load_boston() data = BostonData.data labels = BostonData.target # تعريف النماذج التي سنطبقها model1 = SVR() model2 = DecisionTreeRegressor() model3 = RandomForestRegressor() model = [model1 , model2 , model3] j=1 for m in model: print('result of model number : ' , j ,' for cv value ',n,' is ' , cross_val_score(m, X, y, cv=3)) print('-------------------------------------------------------------------------------------------') j+=1 #result of model number : 1 for cv value 10 is [0.51272653 0.75456596 0.69387067] #------------------------------------------------------------------------------------------- #result of model number : 2 for cv value 10 is [0.41026494 0.64218456 0.54842306] #------------------------------------------------------------------------------------------- #result of model number : 3 for cv value 10 is [0.65818535 0.8420133 0.80363026] #-------------------------------------------------------------------------------------------
- 1 جواب
-
- 1
-

-
عند تعاملك مع القيم المفقودة في واصفة ما (أو مع feature معينة بشكل عام) يجب أن تنتبه إلى طريقة استبدالك للقيم المفقودة فغالباً يقوم الجميع باستبادال القيم nan مثلاً بقيمة 0 وهذا سوف يسبب خطأ لأنه أصبح لديك هنا نوعين من البيانات، وهذا سينتج خطأ لأن الكلاس LabelEncoder سيتوقع منك String بينما قمت بإعطائه أنواع متعددة من البيانات. حل مشكلتك يكون واحدة من الاثنين: 1.في حال لم تستبدل القيم المفقودة بعد: فاجعل القيمة المعوضة لها بين ""، فمثلاً تريد تعويض nan ب 0. # "" لاحظ أنني وضعتها بين fillna('0') 2.في حال قمت فعلاً بتعويضها ب 0 بدون أن تضعها ضمن "". dataframe["Embarked"] = dataframe["Embarked"].astype(str)
- 1 جواب
-
- 1
-

-
هذا سببه أن عمود بياناتك يحوي أنماط مختلفة من البيانات (string و float) بدل أن يحتوي نوع واحد فقط وهو string ولحل هذه المشكلة قم بالتالي: dataframe['column_name'] = dataframe['column_name'].astype(str) وبشكل عام قد يظهر هذا الخطأ أيضاً إذا كانت لديك قيم مفقودة.
- 1 جواب
-
- 1
-

-
كمبرمجين في لغة بايثون نستخدم هذه التعليمة أثناء بناء الكود (Code)، حيث نقوم باستخدامه لينوب عن مقطع برمجي (SubCode) "وليكن A " نريد تركه فارغاً حالياً وتأجيل كتابته إلى وقت آخر. لكن أن نترك المقطع فارغاً هو شيء غير مسموح وينتج عنه خطأ في حال كان ال SubCode هو تابع أو كلاس أو تعبير شرطي أو حلقة، فكما تعلم التعليمة التالية تنتج خطأ إذا لم نعرف شيء ضمن التابع (لايمكن تركه فارغاً) def C(): print("OK") #فارغاً وهذا سينتج خطأ A هنا تركنا التابع def A(): print(5+6) هنا تأتي مهمة التعليمة pass، وعندما يتم تنفيذ هذه التعليمة لايحدث أي شيء ولكن تتجنب أن يتم إيقاف التنفيذ والحصول على error . أي: def C(): print("OK") # هنا لن ينتج خطأ def A(): pass print(5+6) نفس الأمر لو كنت تستخدم حلقة مثلاً: for x in [0, 1, 2]: pass مثال: s = {'d', 'c', 'k', 's'} for v in s: pass # هنا سيمر المترجم على الحلقة ولن يتم تنفيذ أي شيء أي كأنها لاحوي على تعليمات أو في حال قمنا بتعريف Class: class V: pass الخلاصة : نستخدمها عندما نريد تحقيق مقطع برمجي (حلقة أو تابع أو كلاس أو عبارة شرطية ...إلخ) لكن لسبب ما نريد تحقيقها في وقت لاحق، ونرغب بوضعها في الكود فارغة.
- 2 اجابة
-
- 2
-

-
يمكنك ذلك عبر الموديول: sklearn.feature_selection.SelectFromModel الصيغة: sklearn.feature_selection.SelectFromModel(estimator, max_features=None) الوسيط الأول يعبر عن الموديل الذي تريد أن تستخدمه. الوسيط الثاني يعبر عن أكبر عدد تسمح به من ال features. في المثال التالي سأوضح لك الأمر بسهولة: from sklearn.feature_selection import SelectFromModel from sklearn.datasets import load_breast_cancer from sklearn.ensemble import RandomForestClassifier X = load_breast_cancer().data y = load_breast_cancer().target print(X.shape)# (569,30) #لاستخراج أفضل الميزات RandomForestClassifier هنا سنقوم باستخدام الموديل clf = SelectFromModel(estimator=RandomForestClassifier(n_estimators = 20),max_features = None) #على البيانات fitting عمل # والتحويل بعدها X = clf.fit_transform(X, y) #clf.get_support() print(X.shape) # (569,10)
- 1 جواب
-
- 1
-

-
يقوم هذا الصف باختيار أهم ال Features في بياناتك التي تؤثر بقيم ال target اعتماداً على خوارزميتين f_classif أو chi2. يتم استدعاؤه كالتالي: sklearn.feature_selection.SelectKBest ويجب أن نقوم أيضاً باستدعاء خوارزميتي f_classif أو chi2: sklearn.feature_selection.chi2 , f_classif الصيغة: sklearn.feature_selection.SelectKBest(score_func=chi2, k=10) K: افتراضياً 10، يحدد عدد الفيتشرز التي سيحتفظ بها تبعاً لمدى تأثيرها في قيم الخرج، وتقبل عدد صحيح يعبر عن العدد المطلوب، بالإضافة إلى القيمة all التي تستخدم للبحث عن المعلمات (يحتفظ بكل الفيتشرز لكن يستخدم عادةً لمعرفة مدى تأثير كل فيتشر) score_func: تمثل الخوارزمية التي نريد تطبيقها f_classif أو chi2. مثال: # تحميل مايلزم من مكتبات from sklearn.datasets import load_breast_cancer from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import chi2 , f_classif # تحميل الداتا data, target = load_breast_cancer(return_X_y=True) # حجمالداتا قبل التطبيق data.shape #(569, 30) fs = SelectKBest(score_func= chi2 ,k='all') data = fs.fit_transform(data,target) # حجمالداتا بعد تطبيق التحويل data.shape #(569, 10) #لكل ميزة score عرض ال #fs.scores_ ولعرض النتيجة التي حققتها كل feature نستخدم الواصفة scores_. ونستخدم التابع get_support لعرض الفيتشرز التي تم الاحتفاظ بها والتي تم استبعاها.
- 1 جواب
-
- 1
-

-
هذا الخطأ قد يظهر معك في أي نوع من أنواع التقسيم الأخرى التي تندرج تحت فكرة KFolds والسبب فيها أنك تحاول تقسيم البيانات لعدد أكبر من حجم العينات التي لديك. لذا فعدد التقسيمات n_splits يجب أن يكون أكبر أو يساوي عدد العينات التي لديك وفي حالتك هو 3. import numpy as np from sklearn.model_selection import RepeatedKFold X = np.array([[3, 32], [2, 9], [15, 8]]) y = np.array([11, 22, 33]) rkf = RepeatedKFold(n_splits=3, n_repeats=4, random_state=44) for train_index, test_index in rkf.split(X): print("TRAIN:", train_index, "TEST:", test_index) X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index]
- 2 اجابة
-
- 2
-

-
توفر مكتبة Sklearn الكلاس SimpleImputer للتعامل مع القيم المفقودة، من خلال الموديول impute: sklearn.impute.SimpleImputer الصيغة: sklearn.impute.SimpleImputer(missing_values=nan, strategy='mean', fill_value=None,copy=True) strategy:تعني الطريقة التي سيتم التعامل بها مع القيم المفقودة mean , median , most_frequent , constant. mean سيعوض القيمة المفقودة بال mean للقيم المجاورة. most_frequent سيعوضها بالقيمة الأكثر تكراراً. constant بقيمة ثابتة أنت تحددها. median سيعوضها بال median للقيم المجاورة. copy: فيحال ضبطها على true سيتم إنشاء نسخة من البيانات أي لن يكون التعديل على البيانات الأصلية. missing_values: تحديد القيم التي تعتبرها كقيم مفقودة في بياناتك (مثلاً وجود NAN يعني أن القيمة مفقودة أو 0 مثلاً). ويأخذ القيم : "int, float, str, np.nan or None, default="np.nan fill_value: نستخدم هذا الوسيط في حالة استخدمنا استراتيجية constant حيث نسند له القيمة التي نريد الاستبدال بها. افتراضياً None. نستخدم التابع fit للقيام بعملية ال fitting على الداتا (اكتشاف القيم المفقودة و العمليات اللازمة للتعامل مع القيم المفقودة ) نستخدم التابع transform(data) لتطبيق التحويل (استبدال القيم المفقودة) مثال: #استيراد المكتبات اللازمة import numpy as np from sklearn.impute import SimpleImputer # إنشاء داتا بسيطة بقيمة مفقودة data=[[3, 4], [np.nan, 6]] #SimpleImputer تطبيق الكلاس imp = SimpleImputer(missing_values=np.nan, strategy='constant',fill_value=5) #على البيانات fitting عمل imp.fit(data) # تطبيق التحويل data=imp.transform(data) print(data) ''' [[3. 4.] [5. 6.]] '''
- 2 اجابة
-
- 1
-

-
يقوم هذا الصف باختيار أهم ال Features في بياناتك التي تؤثر بقيم ال target اعتماداً على خوارزميتين f_classif أو chi2. يتم استدعاؤه كالتالي: sklearn.feature_selection.GenericUnivariateSelect ويجب أن نقوم أيضاً باستدعاء خوارزميتي f_classif أو chi2: sklearn.feature_selection.chi2 , f_classif الصيغة المبسطة للكلاس: GenericUnivariateSelect(score_func= f_classif, mode= 'k_best', param=n) score_func: تمثل الخوارزمية التي نريد تطبيقها f_classif أو chi2. param: تمثل عدد ال features التي نريد أن نحتفظ بها من ال features الكلية الموجودة في بياناتنا، ويأخذ عدد صحيح يمثل العدد المطلوب، فلو وضعنا 40 فهذا يعني أنه سيتم الاحتفاظ ب 40 فيتشرز (الأكثر أهمية من وجهة نظر الخوارزمية) ويتم استبعاد الباقي. mode: يحدد نمط الاختيار ويكون :{‘k_best’, ‘fpr’, ‘fdr’, ‘fwe’}, default="percentile بحيث k_best يختار أفضل k فيتشر (الفيتشرز التي أعطت أعلى k دقة (score))، أما الباقي فيعتمد على تفاصيل إحصائية في اختيار ال n فيتشرز هي ال false positive rate و false discovery rate و family wise error. ولعرض النتيجة التي حققتها كل feature نستخدم الواصفة scores_. ونستخدم التابع get_support لعرض الفيتشرز التي تم الاحتفاظ بها والتي تم استبعاها. مثال: from sklearn.datasets import load_breast_cancer from sklearn.feature_selection import GenericUnivariateSelect from sklearn.feature_selection import chi2 , f_classif data, target = load_breast_cancer(return_X_y=True) data.shape #(569, 30) fs = GenericUnivariateSelect(chi2, mode='k_best', param=15) data = fs.fit_transform(data,target) data.shape #(569, 15) #لكل ميزة score عرض ال #fs.scores_
- 1 جواب
-
- 1
-

-
يقوم هذا الصف باختيار أهم ال Features في بياناتك التي تؤثر بقيم ال target اعتماداً على خوارزميتين f_classif أو chi2. يتم استدعاؤه كالتالي: sklearn.feature_selection.SelectPercentile ويجب أن نقوم أيضاً باستدعاء خوارزميتي f_classif أو chi2: sklearn.feature_selection.chi2 , f_classif الصيغة: SelectPercentile(score_func = f_classif , percentile=persentage) score_func: تمثل الخوارزمية التي نريد تطبيقها f_classif أو chi2. percentile: تمثل النسبة المئوية التي نريد اختيارها من الداتا، ويأخذ قيم بين ال 0 و ال 100 فمثلاً لو وضعنا 50 فهذا يعني أنه سيتم اختزال حجم الفيتشرز إلى النصف بحيث تكون ال 50% التي سيتم الاحتفاظ بها أهم الفيتشرز والباقي تستبعد لأنها أقل أهمية (أقل تأثيراً على قيم ال target) من وجهة نظر الخوارزمية. ولعرض النتيجة التي حققتها كل feature نستخدم الواصفة scores_. ونستخدم التابع get_support لعرض الفيتشرز التي تم الاحتفاظ بها والتي تم استبعاها. مثال: from sklearn.datasets import load_breast_cancer from sklearn.feature_selection import SelectPercentile from sklearn.feature_selection import chi2 , f_classif data, target = load_breast_cancer(return_X_y=True) data.shape #(569, 30) fs = SelectPercentile(chi2, percentile=50) data = fs.fit_transform(data,target) data.shape #(569, 15) #لكل ميزة score عرض ال #fs.scores_
- 1 جواب
-
- 1
-

-
كما تشير وثيقة Sklearn فإن ال estimator الخاص بهذه الخوارزمية مازال تجريبي (experimental) ولاستخادمها تحتاج إلى تمكين الميزات التجريبية أولاً (experimental features) ويتم ذلك عن طريق الاستدعاء التالي: from sklearn.experimental import enable_hist_gradient_boosting أي يصبح الكود: from sklearn.model_selection import train_test_split from sklearn.experimental import enable_hist_gradient_boosting from sklearn.ensemble import HistGradientBoostingClassifier Data = load_breast_cancer() X = Data.data y = Data.target X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=44, shuffle =True) clf = BaggingClassifier(n_estimators=150, random_state=444) clf.fit(X_train, y_train)
- 1 جواب
-
- 1
-

-
أفضل دوماً أن أبدأ بمقدمة بسيطة عندما يتعلق الأمر بخوارزميات التعليم بدون إشراف، إنها طريقة لعمل تقسيم للبيانات الغير معنونة، unlabeled data وتستخدم في التعلم بلا إشراف وهي مجموعة من خوارزميات التقسيم التي تبني تقسيمات أو مجموعات متداخلة على التوالي ,يتم تمثيل هذا التسلسل الهرمي للعناقيد كشجرة هرمية حيث أن أوراق هذه الشجرة هي عناقيد بعينة واحدة فقط وجذرها هو مجموعة العناقيد كلها حيث يقوم AgglomerativeClustering بعملية التقسيم من أسفل إلى أعلى حيث تبدأ كل عينة في مجموعة خاصة بها ثم يتم دمج المجموعات على التوالي مثل تجمع الطرق . يتم استخدامها عبر الموديول sklearn.cluster. استدعاء المكتبات: from sklearn.cluster import AgglomerativeClustering في البداية قمنا باستدعاء المكتبة التي يوجد فيها هذا الموديل. الشكل العام للموديل: AggModel=AgglomerativeClustering(n_clusters=2, affinity='euclidean', memory=None, linkage='ward',distance_threshold=None,compute_distances=False) الوسيط الأول قيمة صحيحة تمثل عدد العناقيد . الوسيط الثاني affinity المقياس المستخدم لعملية الدمج بين المجموعات ويمكن أن يكون المسافة الأقليدية أو مسافة منهاتن أو المحسوبة سابقا أي يكون هنالك مصفوفة تحوي قيم المسافات. الوسيط الثالث memory بالحالة الافتراضية يتم استخدام الذاكرة cacah لحساب الشجرة الهرمية. الوسيط الرابع linkage معيار الربط أو الوصل بين المجموعات حيث يحدد هذا المعيار المسافة التي يجب استخدامها بين مجموعتين حيث يتم دمج كل مجموعتين تمتلكان أقل مسافه وهكذا.. الوسيط الخامس distance_threshold عتبة المسافة تكون مثل خط أفقي على أوراق الشجرة كل مجموعة من الأوراق فوقها تعتبر عنقود أي مجموعة تحتها لا يعتبر .. الوسيط السادس compute_distances يستخدم لحساب المسافات بين المجموعات.. طبعا قمت بشرح الشكل العام للموديل ولكن عن طريقة استخدامه سهل جدا فقط بعد أن تقوم بتقسيم الداتا إلى X_train,X_test تسطيع كتابة الأتي لعملية التدريب الشكل العام للموديل: AggModel=AgglomerativeClustering(n_clusters=2, affinity='euclidean', memory=None, linkage='ward',distance_threshold=None,compute_distances=False) AP.fit(X_train) حيث الدالة fit يوجد ضمنها جميع العمليات الداخلية لعملية التدريب . يوجد دالة أخرى تستخدم لغرض التنبؤ، نستطيع فيها حساب قيم التقسيم على التدريب والاختبار عن طريق التابع fit_predict ويكون وفق الشكل: # طباعة الخرج على الاختبار والتدريب y_pred_train = AP.fit_predict(X_train) y_pred_test = AP.fit_predict(X_test) print('AP Train data are : ' ,y_pred_train) print('AP Test data are : ' ,y_pred_test) لنأخذ مثال يوضح الموديل : from sklearn.cluster import AgglomerativeClustering import numpy as np تعين داتا دخل مزيفة X = np.array([[0, 4], [3, 5], [1, 1], [2, 3], [5, 5], [4, 2]]) بناء الموديل Agg = AgglomerativeClustering() Agg.fit(X) طباعة تصنيف العينه Agg.fit_predict([[0, 0], [4, 4]]) النتيجة array([1, 0], dtype=int64)
- 1 جواب
-
- 1
-

-
قبل أن نحل المشكلة هذه، حبذا أن ننوه إلى أن الأخطاء من توع ImportError تحدث غالباً عندما تستعمل خاصية أو أمر ما موجود في نسخ قديمة ولم يعد موجود في النسخ الحديثة أو العكس، أو يحدث إذا حاولت استخدام مكتبة لم تقم بتثبيتها . وحل هذه المشاكل غالباً يكون إما بتحديث المكتبة أو تثبيتها في حال لم تكن مثبتة. دعنا نعود لمشكلتك: المشكلة لديك هي حالة مشابهة لما ذكرناه والسبب أنه في النسخ السابقة ل Sklearn كان التابع train_test_split موجوداً في الموديول cross_validation، لكن في النسخ الحديثة تم نقله إلى الموديول model_selection. أي أن الإصدار الذي لديك من sklearn يجب تحديثه. وبالتالي لحل مشكلتك إما أن تستدعيه من الموديول الصحيح أي: from sklearn.cross_validation import train_test_split أو أن تقوم بتحديث النسخة التي لديك. عن طريق مدير الحزم كوندا في بيئة أناكوندا: conda update scikit-learn # أو conda install scikit-learn=0.24.2 أو باستخدام pip: pip install -U scikit-learn
- 2 اجابة
-
- 1
-

-
الكلاسيفير المفضل لدي. يمكنك استخدامه عن طريق الموديول: sklearn.ensemble الصيغة العامة: sklearn.ensemble.ExtraTreesClassifier(n_estimators=100, criterion='gini', max_depth=None, min_samples_split=2, min_samples_leaf=1, max_features='auto', max_leaf_nodes=None, bootstrap=False, oob_score=False,n_jobs=None, random_state=None, verbose=0, warm_start=False,ccp_alpha=0.0) n_estimators : عدد أشجار القرار المستخدمة. default=100 criterion: الأسلوب الرياضي للمعالجة وتكون {“gini”, “entropy”}, 'default='gini max_depth : عمق الأشجار. min_samples_split:الحد الأدنى لعدد العينات المطلوبة لتقسيم عقدة داخلية. int , default=2. min_samples_leaf: الحد الأدنى لعدد العينات المطلوبة في العقدة التي تمثل الاوراق. default=1. max_features:العدد المناسب من الفيتشرز التي يتم احتسابها {“auto”, “sqrt”, “log2”}. في حال auto: max_features=sqrt(n_features). sqrt: ax_features=sqrt(n_features). log2: max_features=log2(n_features). None: max_features=n_features. إذا وضعت قيمة float: max_features=int(max_features * n_features) قيمة int: سيتم أخذ ال features عند كل تقسيمة ك max_features. bootstrap: لتحديد فيما إذا كان سيتم استخدام عينات ال bootstrap عند بناء الأشجار. في حال ضبطها على true سيتم استخدام كامل البيانات لبناء كل شجرة. افتراضياً تكون False. oob_score: لتحديد فيما إذا كان سيتم استخدام عينات out-of-bag لتقدير قيمة التعميم "generalization score". ويجب أن تكون bootstrap مضبوطة على True لاستخدامها. n_jobs: عدد المهام التي يتم تنفيذها بالتوازي. -1 للتنفيذ بأقصى سرعة ممكنة. random_state: يتحكم بعملية التقسيم افتراضياً يكون None. verbose: لعرض التفاصيل التي تحدث في التدريب. افاراضياً 0 أي لايظهر شيء، أما وضع أي قيمة أكبر من الصفر سيعرض التفاصيل int. ccp_alpha: معامل تعقيد يستخدم لتقليل التكلفة الزمانية والمكانية. non-negative float, default=0.0 التوابع: fit(data): للقيام بعملية التدريب. predict(data): لتوقع القيم. score(data): لتقييم كفاءة النموذج. ()get_params :لايجاد مقدار الدقة predict_proba(data) : لعمل التوقع أيضاً لكن هنا سيخرج الفيمة الاحتمالية(أي لن يتم القصر على عتبة) apply(data): ياتي لك بقيمة الورقة المحسوبة. ()get_n_leaves: يرد عدد الأوراق. ()get_depth: يرد عمق الشجرة. ال attributtes: classes_: لعرض ال labels التي وجدها. n_classes_: عددها. n_outputs_: عدد المرخرجات الناتجة عن عملية ال fitting. estimators_: عرض معلومات عن كل الأشجار التي تم تشكيلها. base_estimator_:عرض معلومات الشجرة الأساسية. n_features_: عدد الفيتشرز. مثال: from sklearn.ensemble import ExtraTreesClassifier from sklearn.model_selection import train_test_split from sklearn.datasets import load_breast_cancer # تحميل البيانات Data = load_breast_cancer() X = Data.data y = Data.target # تقسيم البيانات إلى عينات تدريب واختبار X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=44, shuffle =True) # ExtraTreesClassifier تطبيق clf = ExtraTreesClassifier(n_estimators=150, random_state=444) clf.fit(X_train, y_train) # النتائج print('ExtraTreesClassifier Train Score is : ' , clf.score(X_train, y_train)) # ExtraTreesClassifier Train Score is : 1.0 print('ExtraTreesClassifier Test Score is : ' , clf.score(X_test, y_test)) # ExtraTreesClassifier Test Score is : 0.9736842105263158
- 1 جواب
-
- 1
-

-
يمكنك القيام بذلك عن طريق الموديول ensemble في مكتبة Sklearn. BaggingClassifier(base_estimator=None, n_estimators=10,max_samples=1.0, bootstrap=True, oob_score=False, n_jobs=None, random_state=None, verbose=0) base_estimator: ال estimator الأساسي الذي سيتم استخدامه لعمل fitting على مجموعات فرعية عشوائية من مجموعة البيانات. افتراضياً يكون DecisionTreeClassifier. أي إذا وضعت None. (يقبل object) n_estimators: عدد ال estimator التي تريد أن يتم تطبيقها. افتراضياً 10. bootstrap: لتحديد فيما ما إذا كان سيتم سحب العينات مع الاستبدال. إذا كان Flase، سيتم إجراء أخذ العينات بدون استبدال. oob_score: لتحديد فيما إذا كان سيتم استخدام عينات out-of-bag لتقدير قيمة التعميم "generalization score". ويجب أن تكون bootstrap مضبوطة على True لاستخدامها. n_jobs: عدد المهام التي يتم تنفيذها بالتوازي. -1 للتنفيذ بأقصى سرعة ممكنة. random_state: يتحكم بعملية التقسيم افتراضياً يكون None. verbose: يتحكم بال verbosity أثناء التدريب والتنبؤ. التوابع: fit(data): للقيام بعملية التدريب. predict(data): لتوقع القيم. score(data): لتقييم كفاءة النموذج. predict_proba(data) : لعمل التوقع أيضاً لكن هنا سيخرج الفيمة الاحتمالية(أي لن يتم القصر على عتبة) apply(data): ياتي لك بقيمة الورقة المحسوبة. ()get_n_leaves: يرد عدد الأوراق. ()get_depth: يرد عمق الشجرة. ال attributtes: classes_: لعرض ال labels التي وجدها. n_outputs_: عدد المرخرجات الناتجة عن عملية ال fitting. estimators_: عرض معلومات عن كل ال estimator التي تم تشكيلها. base_estimator_:عرض معلومات ال estimator الأساسية. n_features_: عدد الفيتشرز. مثال: from sklearn.ensemble import BaggingClassifier from sklearn.model_selection import train_test_split from sklearn.metrics import confusion_matrix from sklearn.datasets import load_breast_cancer import seaborn as sns import matplotlib.pyplot as plt # تحميل البيانات Data = load_breast_cancer() X = Data.data y = Data.target # تقسيم البيانات إلى عينات تدريب واختبار X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=44, shuffle =True) # BaggingClassifier تطبيق clf = BaggingClassifier(n_estimators=150, random_state=444) clf.fit(X_train, y_train) # النتائج print('BaggingClassifier Train Score is : ' , clf.score(X_train, y_train)) # BaggingClassifier Train Score is : 1.0 print('BaggingClassifier Test Score is : ' , clf.score(X_test, y_test)) # BaggingClassifier Test Score is : 0.0.9649122807017544 # عرض مصفوفة التشتت c = confusion_matrix(y_test, clf.predict(X_test)) print('Confusion Matrix is : \n', c) #لرسم المصفوفة sns.heatmap(c, center = True) plt.show()
- 2 اجابة
-
- 1
-

-
هذا خطأ عام، وقد يظهر لك عند استخدامك لأي خوارزمية أخرى أوشبكة عصبية ويحدث عند محاولتك قياس كفاءة نموذج باستخدام معيار f1-score مع مسائل التصنيف المتعدد. الخطأ في السطر التالي: f1_score(y_test,t.predict(x_test)) إن مجموعة البيانات الشهيرة MINST تشكل مسألة تصنيف متعدد، لذا لايمكن استخدام معيار f1-score معها كما في الشكل الذي أرفقته. الشكل العام للتابع: f1_score(y_true, y_pred,average='binary') أي افتراضياً يكون ال average تم تعيينه على binary (أي يقيس الكفاءة لمسألة تصنيف ثنائي)، لكن مسألتك هي مسألة تصنيف متعدد وبالتالي لايصلح استخدامه هنا. لحل المشكلة نستخدم أحد المعاملات ['None, 'micro', 'macro', 'weighted]. طبعاً يمكنك استخدام أي منها ولا يعطونك نتائج متفاوتة (غالباً تكون الفروقات بقيم مهملة) وبشكل عام فإن [None, 'micro', 'macro', 'weighted'] يمكنك استخدامها في حالة التصنيف المتعدد والثنائي. الحل: import numpy as np from tensorflow.keras.datasets import mnist from sklearn.linear_model import LogisticRegression from sklearn.pipeline import Pipeline from tensorflow.keras.utils import to_categorical from sklearn.metrics import f1_score,precision_score,recall_score,accuracy_score,log_loss (x_train, y_train),(x_test, y_test) = mnist.load_data() x_train.shape image_size = x_train.shape[1] input_size = image_size * image_size x_train = np.reshape(x_train, [-1, input_size])/ 255 x_test = np.reshape(x_test, [-1, input_size]) / 255 t =LogisticRegression() t.fit(x_train, y_train) f1_score(y_test,t.predict(x_test),average='micro') #0.9258 # أو #f1_score(y_test,t.predict(x_test),average='macro') #0.9247 # أو #f1_score(y_test,t.predict(x_test),average=None) weighted: استخدمها إذا أردت أن تكون قيمة f1 معتمدة على عدد عينات كل صنف أيضاً . macro: تأخذ متوسطات ال f1 لكل كلاس. micro: نفس مبدأ accuracy. None: تعطيك ال f1 لكل فئة .
- 2 اجابة
-
- 2
-

-
عندما تستخدم import module مثلاً import sklearn تكون قد قمت باستيراد كامل المكتبة (بكل الموديول التي فيها وبالتالي بكل الكلاسات وكأنك قمت بتعريف قبضة يمكنها مسك أي شيء داخل المكتبة) وبالتالي يصبح بإمكانك استداعاء أي شيء منها عن طريق ذكر اسم المكتبة ثم اسم الموديول ثم اسم الكلاس ووضع نقطة بينهم. مثال: #seaborn قمت باستداعاء المكتبة import seaborn #heatmap من خلال اسم المكتبة أستطيع الوصول إلى الدالة المعرفة بداخلها التي تسمى seaborn.heatmap(c, center = True) # ويمكننا اعطاء اسم محتصر للمكتبة كالتالي import seaborn as sea sea.heatmap(c, center = True) مثال: #sklearn قمت باستيراد مكتبة import sklearn #metrics الموجود داخل الموديول confusion_matrix أريد الآن استخدام الكلاس c = sklearn.metrics.confusion_matrix(y_test, clf.predict(X_test)) # لاحظ كيف كتبنا اسم المكتبة ثم الموديول ثم الكلاس أما في حالة استخدمنا الطريقة الثانية فنكون قد قمنا باستيراد شيء محدد من المكتبة أو الموديول ولانكون قد استوردنا غيره أي وكأنك عرفت قبضة على جزء محدد من المكتبة. مثال #sklearn الموجود في مكتبة metrics من الموديول confusion_matrix هنا قمنا باستيراد ال from sklearn.metrics import confusion_matrix c = confusion_matrix(y_test, clf.predict(X_test)) #matplotlibالموجود ضمن المكتبة pyplot الآن مثال آخر حيث سنستورد كل مابداخل الموديول from matplotlib import pyplot # pyplot وبالتالي أصبح بإمكانك الوصول لكل مايداخل الموديول #show مثلاً أريد الوصول للتابع pyplot.show() :
- 2 اجابة
-
- 2
-

-
نستخدم الدالة open التي تعيد لنا كائن من النوع file أي file object ثم في حالة القراءة لانمرر أي شيء للدالة open أما إذا أردنا التعديل (الإضافة في نهاية الملف) نمرر لها "a" وإذا أردنا الكتابة فوق الملف الموجود نمرر "w" ثم نستدعي الدالة write ونمرر لها مانريد أن يتم إضافته كالتالي: # الإضافة على الملف file = open("D:/r.txt", "a") file.write(" add line ") file.close() # نعيد فتح الملف لنتأكد من الإضافة file = open("D:/r.txt", "r") print(file.read()) # w نجرب الآن ال file = open("D:/r.txt", "w") file.write("say hi") file.close() # نعيد فتح الملف لنتأكد من الإضافة file = open("D:/r.txt", "r") print(file.read()) ولاننسى إغلاق الملف بعد الانتهاء باستخدام الدالة close.
- 2 اجابة
-
- 1
-

-
هذا الخطأ قد يظهر لك في أي خوارزمية أخرى قد تستخدمها. المشكلة في أنك تقوم بترميز قيم ال labels باستخدام ال One-Hot Encoding (التابع to_categorical) فتحدث المشكلة في التابع fit أي عندما يبدأ التدريب. صحيح أننا نستخدم عادةً التابع to_categorical لترميز ال labels في مسائل التصنيف المتعدد لكن في الخوارزميات المعرفة في مكتبة Sklearn لانستخدمه لأن الخوارزميات فيها لاتتعامل مع هذا الشكل من البيانات. إن شكل التابع fit في مكتبة Sklearn كالتالي: fit(X, Y) بحيث أن y هي مصفوفة من الشكل (,n_sampels) أي مصفوفة أحادية الأبعاد (1D-array). وعند استخدامك لترميز to_categorical سوف يتحول شكل البيانات إلى (n_sampels,voc) بحيث voc هي عدد ال labels الموجودة. وهذا الشكل لايتطابق مع شكل البيانات الذي تتعامل معه fit. لذا يجب أن لانقوم باستخدام هذا الترميز وأن نجعل ال labels ضمن مصفوفة 1D: حل المشكلة: import numpy as np from sklearn.naive_bayes import MultinomialNB from tensorflow.keras.utils import to_categorical from tensorflow.keras.datasets import mnist from sklearn.metrics import accuracy_score (X, Y),(Xtest, Ytest) = mnist.load_data() Y=Y.reshape(-1,) Ytest=Ytest.reshape(-1,) X = np.reshape(X, [-1, X.shape[1]*X.shape[1]]) Xtest = np.reshape(Xtest, [-1, input_size]) M =MultinomialNB() M.fit(X, Y) accuracy_score(Ytest,M.predict(Xtest)) # 0.83
- 2 اجابة
-
- 1
-

-
خوارزمية تعتمد على التصويت بين عدة خوارزميات، بحيث تحدد لها عدة خوارزميات توقع وكل خوارزمية ستقوم بعمل fitting على البيانات ثم إجراء مايسمى "Voting" لانتخاب النتيجة الأفضل اعتماداً على الخوارزميات المستخدمة. (تشبه VotingClassifier لكن هنا لمهمة توقع). يمكنك استخادمها عبر الموديول: sklearn.ensemble.VotingRegressor(estimators, weights=None, n_jobs=None) الوسيط الأول نحدد فيه خوارزميات التوقع التي نريد استخدامها وتقبل list من ال tuble بحيث كل tuble عبارة عن قيمة أولى str تمثل اسم اختياري للخوارزمية وقيمة ثانية تمثل الكلاس (الخوارزمية) "موضحة في المثال". الوسيط weights: تحديد ماهي الأوزان في التصويت لكل خوارزمية. ويأخذ مصفوفة من الأوزان (,n_classifier,) قد تكون القيم int أو float لامشكلة. n_jobs: عدد المهام التي يتم تنفيذها على التوازي. نضع -1 لأقصى قدر ممكن(زيادة سرعة التنفيذ). ال attributes: estimators_ : معلومات عن الخوارزميات المستخدمة. التوابع: fit(data) للقيام بعملية التدريب. predict(data) للقيام بعملية توقع قيمة عينة. score(data) لإيجاد كفاءة النموذج. مثال: قمنا هنا باستخدام خوارزميتي توقع RandomForest+LinearRegression import numpy as np from sklearn.ensemble import RandomForestRegressor from sklearn.linear_model import LinearRegression from sklearn.ensemble import VotingRegressor reg1 = LinearRegression() reg2 = RandomForestRegressor(n_estimators=7, random_state=2021) # تشكيل بيانات بقيم عشوائية X = np.array([[3, 1], [32, 45], [53, 2], [5, 6]]) y = np.array([2, 6, 12, 20]) #VotingRegressor تعريف reg = VotingRegressor([('reg1', reg1), ('reg2', reg2)]) print(reg.fit(X, y).predict(X)) # [ 9.68531265 8.55106081 11.01620204 15.17599594] print(reg.score(X, y)) # 0.5118977425789101
- 1 جواب
-
- 1
-