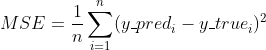ريم المهدي
الأعضاء-
المساهمات
127 -
تاريخ الانضمام
-
تاريخ آخر زيارة
نوع المحتوى
ريادة الأعمال
البرمجة
التصميم
DevOps
التسويق والمبيعات
العمل الحر
البرامج والتطبيقات
آخر التحديثات
قصص نجاح
أسئلة وأجوبة
كتب
دورات
كل منشورات العضو ريم المهدي
-
لقد وجدت نوع من انواع GANs يعمل على إنشاء صور و تصنيفات وهو ما يسمي ب Conditional Generative Adversarial Netowrks او cGANs، البرنامج السابق يستخدم فقط shallow GANs وهي أنواع الخوارزميات التي تختص فقط بإنشاء صور جديدة. Answering this in case anyone else falls into the same trap
- 1 جواب
-
- 3
-

-
كيف يمكن أن أحصل على ال labels للصور المنشأة بواسطة GANs، مثلاً عند إستخدام GANs لزيادة عدد صور MNIST dataset و الحصول على الصور، كيف أعرف ال label لكل صورة دون أن أقوم بذلك يدوياً. import os import numpy as np import matplotlib.pyplot as plt from tqdm import tqdm from keras.layers import Input from keras.models import Model, Sequential from keras.layers.core import Dense, Dropout from keras.layers.advanced_activations import LeakyReLU from keras.datasets import mnist from keras.optimizers import Adam from keras import initializers # Let Keras know that we are using tensorflow as our backend engine os.environ["KERAS_BACKEND"] = "tensorflow" # To make sure that we can reproduce the experiment and get the same results np.random.seed(10) # The dimension of our random noise vector. random_dim = 100 def load_minst_data(): # load the data (x_train, y_train), (x_test, y_test) = mnist.load_data() # normalize our inputs to be in the range[-1, 1] x_train = (x_train.astype(np.float32) - 127.5)/127.5 # convert x_train with a shape of (60000, 28, 28) to (60000, 784) so we have # 784 columns per row x_train = x_train.reshape(60000, 784) return (x_train, y_train, x_test, y_test) # You will use the Adam optimizer def get_optimizer(): return Adam(lr=0.0002, beta_1=0.5) def get_generator(optimizer): generator = Sequential() generator.add(Dense(256, input_dim=random_dim, kernel_initializer=initializers.RandomNormal(stddev=0.02))) generator.add(LeakyReLU(0.2)) generator.add(Dense(512)) generator.add(LeakyReLU(0.2)) generator.add(Dense(1024)) generator.add(LeakyReLU(0.2)) generator.add(Dense(784, activation='tanh')) generator.compile(loss='binary_crossentropy', optimizer=optimizer) return generator def get_discriminator(optimizer): discriminator = Sequential() discriminator.add(Dense(1024, input_dim=784, kernel_initializer=initializers.RandomNormal(stddev=0.02))) discriminator.add(LeakyReLU(0.2)) discriminator.add(Dropout(0.3)) discriminator.add(Dense(512)) discriminator.add(LeakyReLU(0.2)) discriminator.add(Dropout(0.3)) discriminator.add(Dense(256)) discriminator.add(LeakyReLU(0.2)) discriminator.add(Dropout(0.3)) discriminator.add(Dense(1, activation='sigmoid')) discriminator.compile(loss='binary_crossentropy', optimizer=optimizer) return discriminator def get_gan_network(discriminator, random_dim, generator, optimizer): # We initially set trainable to False since we only want to train either the # generator or discriminator at a time discriminator.trainable = False # gan input (noise) will be 100-dimensional vectors gan_input = Input(shape=(random_dim,)) # the output of the generator (an image) x = generator(gan_input) # get the output of the discriminator (probability if the image is real or not) gan_output = discriminator(x) gan = Model(inputs=gan_input, outputs=gan_output) gan.compile(loss='binary_crossentropy', optimizer=optimizer) return gan # Create a wall of generated MNIST images def plot_generated_images(epoch, generator, examples=100, dim=(10, 10), figsize=(10, 10)): noise = np.random.normal(0, 1, size=[examples, random_dim]) generated_images = generator.predict(noise) generated_images = generated_images.reshape(examples, 28, 28) plt.figure(figsize=figsize) for i in range(generated_images.shape[0]): plt.subplot(dim[0], dim[1], i+1) plt.imshow(generated_images[i], interpolation='nearest', cmap='gray_r') plt.axis('off') plt.tight_layout() plt.savefig('gan_generated_image_epoch_%d.png' % epoch) def train(epochs=1, batch_size=128): # Get the training and testing data x_train, y_train, x_test, y_test = load_minst_data() # Split the training data into batches of size 128 batch_count = x_train.shape[0] / batch_size # Build our GAN netowrk adam = get_optimizer() generator = get_generator(adam) discriminator = get_discriminator(adam) gan = get_gan_network(discriminator, random_dim, generator, adam) for e in xrange(1, epochs+1): print '-'*15, 'Epoch %d' % e, '-'*15 for _ in tqdm(xrange(batch_count)): # Get a random set of input noise and images noise = np.random.normal(0, 1, size=[batch_size, random_dim]) image_batch = x_train[np.random.randint(0, x_train.shape[0], size=batch_size)] # Generate fake MNIST images generated_images = generator.predict(noise) X = np.concatenate([image_batch, generated_images]) # Labels for generated and real data y_dis = np.zeros(2*batch_size) # One-sided label smoothing y_dis[:batch_size] = 0.9 # Train discriminator discriminator.trainable = True discriminator.train_on_batch(X, y_dis) # Train generator noise = np.random.normal(0, 1, size=[batch_size, random_dim]) y_gen = np.ones(batch_size) discriminator.trainable = False gan.train_on_batch(noise, y_gen) if e == 1 or e % 20 == 0: plot_generated_images(e, generator) if __name__ == '__main__': train(400, 128) البرنامج أعلاه يقوم بإنشاء الصور بالصورة المطلوبة لكن كيف أستخرج الlabels?
-
قيمة vocab_size أو max_words أو num_words تشير في طبقة ال embedding إلى عدد الكلمات الكلي التي تم تحويلها إلى قيم رقمية و الذي يجب أن يساوي maximum integer index + 1. لاحظ شكل الطبقة: tf.keras.layers.Embedding( input_dim, output_dim, embeddings_initializer="uniform", embeddings_regularizer=None, activity_regularizer=None, embeddings_constraint=None, mask_zero=False, input_length=None, **kwargs ) والان لنأخذ مثال: from numpy import array from keras.preprocessing.text import one_hot from keras.preprocessing.sequence import pad_sequences from keras.models import Sequential from keras.layers import Dense from keras.layers import Flatten from keras.layers.embeddings import Embedding # تعريف المدخلات كملفات تحتوي على كلمات docs = ['Well done!', 'Good work', 'Great effort', 'nice work', 'Excellent!', 'Weak', 'Poor effort!', 'not good', 'poor work', 'Could have done better.'] # تحديد صنف لكل ملف من الملفات السابقة labels = array([1,1,1,1,1,0,0,0,0,0]) # هنا قمنا بتحديد القيمة 50 كطول للبيانات المدخلة، بالتالي أي كلمة يتم تحويلها لقيمة رقمية مقابلة لها # بأي حال من الأحوال يجب أن تكون القيمة أعلى من العدد الكلي للكلمات المختلفة حتى لا يحصل تصادم vocab_size = 50 encoded_docs = [one_hot(d, vocab_size) for d in docs] print(encoded_docs) # كل ملف يحتوي على كلمات أقل من العدد الذي تم تحديده هنا 4 يتم إضافة أصفار فيه لتصبح كل المدخلات من نفس الطول 4 max_length = 4 padded_docs = pad_sequences(encoded_docs, maxlen=max_length, padding='post') print(padded_docs) # تعريف النموذج model = Sequential() model.add(Embedding(vocab_size, 8, input_length=max_length)) model.add(Flatten()) model.add(Dense(1, activation='sigmoid')) # تنفيذ النموذج model.compile(optimizer='adam', loss='binary_crossentropy', metrics=['accuracy']) # تلخيص النموذج print(model.summary()) # تدريب النموذج model.fit(padded_docs, labels, epochs=50, verbose=0) # تقييم النموذج loss, accuracy = model.evaluate(padded_docs, labels, verbose=0) print('Accuracy: %f' % (accuracy*100)) لاحظ أننا قمنا بتمرير vocab_size إلي طبقة التضمين embedding layer بنفس الطول الذي تم تحديده مسبقاً لترميز البيانات، بالتالي يوجد تطابق ولا ينتج الخطأ السابق.
-
سأقوم بنصيحتك من وجهة نظر عملية: هناك طريقتين للتعلم، من الأعلى للأسفل و العكس، أي الإبتداء من الجبر، الإحصاء و الإحتمالات و بالإضافة للخوارزميات و هيكلية البيانات، و من ثم يمكنك الإطلاع على تعلم الألة و علم البيانات. أو يمكنك الإبتداء من تعلم الألة، علم البيانات و من ثم محاولة فهم كل خوارزمية من خوارزميات تعلم الألة و طريقة عملها، و في التعامل مع البيانات، يمكن اللجوء لهيكلية البيانات، إذ أنها تعطيك الطريقة الأمثل من حيث الوقت و مساحة الذاكرة و غيرها من الخصائص التي تجعلك تختار طريقة عن غيرها. في كل الأحوال هناك عدد من الأشياء يمكنك أخذها بعين الإعتبار لتحديد الطريقة المناسبة لك: إن كنت تركز على إنشاء تطبيقات صغيرة فقط للتعلم يمكنك التعامل مباشرة مع الدوال الجاهزة في علم البيانات وهي التي تقوم بكل المهمات الصعبة مثل تحويل البيانات إلى مستوى أعلى kernel move the data to higher dimension. بالطريقة السابقة يمكنك إنشاء تطبيقات مختلفة مثل التعرف على الصور و توقع القيم المستقبلية لأسعار الأسهم، تقسيم العملاء إلى فئات و غيرها. الشركات المتوسطة و الصغيرة يركزون على إنتاج تطبيقات أكثر من جودتها بالتالي الطريقة السابقة تنفع في هذه الحالة (بالضرورة لا أعني التعميم فقد يوجد بعض الجهات التي تركز على جودة البرامج المقدمة). الشركات الكبيرة تركز على منهجية و طريقة الحل، و تبحث على حلول مستدامة و بالتالي يهمها تقليل التكلفة الناتجة من زمن التنفيذ و المساحة التخزينية، بالأخص في حالة التعامل مع البيانات الضخمة. في حال أردت التعلم للمواصلة في المجال الأكاديمي أنصحك بالبدء من الأساسيات ثم المواصلة، لأنك في هذه الحالة تحاول إيجاد افضل الحلول و ليس أي حل يعمل. في حال كنت تريد عمل تطبيقات متوازنة أو موزعة أنصحك بالبدء بالأساسيات لانها ستساعدك في حال التوسع او إنشاء تطبيقات على خدمات سحابية. في كل الأحوال الخيار يعتمد على ماذا تريد و الطريقة الأنسب لتعملك أنت شخصياً، كل شخص سيعطي الإجابة من وجهة نظره، لا توجد إجابة صحيحة أو خطأ أو إجابة مثالية، هي فقط عرض للخبرات و أنت من يحدد ما الذي سيفيدك. أتمنى أن تكون قد أستفدت.
- 8 اجابة
-
- 3
-

-
الطريقتين متشابهتين بصورة كبيرة لكن localStorage لديها زمن إنتهاء مختلف. sessionStorage يمكن الوصول لمحتوياتها طالما كانت النافذة التي تم إنشاء الجلسة بها مفتوحة، localStorage تكون الجلسة مفتوحة طالما لم تمسحها. مثلاً، في حال كنت تريد حفظ إسم المستخدم و كلمة المرور فإن إستخدام sessionStorage أفضل من إستخدام localStorage و ذلك لأغراض أمنية، أو كان المستخدم يريد إستخدام البرنامج بعد مدة من الزمن. وهي ايضا الدالة التي يمكن إستخدامها في حال كنت تريد حفظ حالة ضبط المستخدم في جهازه. بصورة عامة، localStorage تستخدم في حال كنت تريد حفظ البيانات لمدة أطول و sessionStorage في حال كان الحفظ مؤقت.
-
يجب أن تنتبه أن df.iloc, df.loc, df.at يتم تنفيذهم مع حالة كان index بإستخدام الكلمات كما في المثال او الأرقام، df.iloc تعمل فقط في حال كان integer index. أما df.loc و df.at يمكن أن يعملان في حالة كنت تريد الإضافة بإستخدام إسم العمود مع integer index. في حالة كان index غير معرف فإن كل من df.loc , df.at يقومان بالإضافة في dataframe بصورة مباشرة و لكن df.iloc تقوم بإنشاء خطأ، لاحظ المثال التالي: import numpy as np, pandas as pd df = pd.DataFrame(index=np.arange(3), columns=['x','y','z']) df['x'] = ['A','B','C'] df.at[2,'y'] = 400 # الأسطر و الأعمدة غير موجودة في البيانات الأساسية بالتالي يتم إضافتهم df.at['D','w'] = 9000 df.loc['E','q'] = 499 # هذه الطريقة تقوم بإستخدام عبارة شرطية لفلترة البيانات و من قم الإضافة df.at[df1['x']=='B', 'y'] = 10000 df.loc[df1['x']=='B', ['z','w']] = 10000 # إضافة قيم بإستخدام index df.iloc[[1,2,4], 2] = 9999 df.loc[[0,'D','E'],'w'] = 7500 df.at[[0,2,"D"],'x'] = 10 df.at[:, ['y', 'w']] = 8000 df
- 4 اجابة
-
- 1
-

-
بالاضافة لما شرحه @Ali Haidar Ahmad فإن عدد الطبقات و عدد الوحدات فيها يعتمد بصورة مباشرة على المدخلات و المجال الذي تريد تطبيق شبكة LSTM, GRU, RNN فيه. العدد الأمثل للوحدات hidden units هو أن تكون أقل من العدد المدخل، في حال كان لديك عدد كبير من المدخلات يمكن إستخدام عدد أكبر من hidden units و لكن في بعض الأحيان فقط 2 تكفى. لاحظ شكل LSTM موضحة: هنا ترمز num_units لعدد الوحدات الخفية في الشبكة، وهي تعني المدخل لكل خلية في الشبكة عبارة عن حجم الدفعة batch_size مضروب في عدد الوحدات num_units. العدد 128 ليس قيمة ثابتة، لكن بصورة عامة يمكن أن تبدأ من الطبقة الأولى و تضع فيها عدد وح\ات قريب من عدد المدخلات و يمكنك زيادة العدد و نقصانه تدريجياً حسب القيمة المطلوبة في الخرج.
-
سأقوم بإعطاء أمثلة على الحلقات بإستخدام بايثون. مثال لطباعة أيام الأسبوع: # تحتوي على 7 عناصر days هنا قمنا بتعريف مصفوفة إسمها days = ['Monday', 'Tuesday', 'Wednesday', 'Thursday', 'Friday', 'Saturday', 'Sunday'] # بعدها سيتم عرض قيمته ,day في المتغير days هنا في كل دورة سيتم تخزين قيمة عنصر جديد من عناصر المصفوفة for day in days: print(day) الناتج يكون: Monday Tuesday Wednesday Thursday Friday Saturday Sunday حلقة لطباعة الأعداد من 1 إلى 6 بإستخدام دالة range: # بعدها سيتم عرضه .n هنا قمنا بإنشاء سلسلة من الأرقام الموجودة بين 1 إلى 5. في كل دورة في الحلقة سيتم جلب رقم من هذه السلسلة و تخزينه في المتغير for n in range(1, 6, 1): print(n) الناتج يكون: 1 2 3 4 5 مثال على حلقة while لطباعة جميع القيم بين 1 و 10 بإستخدام متغير i : i = 1 #إنشاء حلقة while تظل تنفذ الأوامر الموضوعة فيها طالما أن قيمة العداد i لا تزال أصغر أو تساوي 10. while i <= 10: print(i) i += 1 والناتج يكون: 1 2 3 4 5 6 7 8 9 10
-
يمكنك ذلك عن طريق str.contains والتي يمكن إستخدامها ل substring search و ايضاً regex based search (والذي يرمز لل regular expression) و هو طريقة البحث by default وقد تأخذ عملية البحث وقت أطول به إذا لم يتم إلغائه في دالة البحث، لاحظ فرق زمن التنفيذ: import pandas as pd df = pd.DataFrame({'col':['cat','hat','bat','dog','ant']}) df1 = pd.concat([df] * 1000, ignore_index=True) %timeit df1[df1['col'].str.contains('og|at')] %timeit df1[df1['col'].str.contains('og|at', regex=False)] أو يمكننا إستخدام دالة find في مكتبة numpy : import numpy as np import pandas as pd df = pd.DataFrame({'col':['cat','hat','bat','dog','ant']}) df[np.char.find(df['col'].values.astype(str), 'at') & np.char.find(df['col'].values.astype(str), 'og') > -1] أو عن طريق دالة query للdf: df.query('col.str.contains("og|at")', engine='python') كما يمكن ذلك عن طريق الدالة vectorize في numpy: import numpy as np import pandas as pd df = pd.DataFrame({'col':['cat','hat','bat','dog','ant']}) f = np.vectorize(lambda words, w: w in words) df[f(df['col'], 'og') | f(df['col'], 'at')]
- 4 اجابة
-
- 1
-

-
يوجد عدد من الخيارات، منها أن يتم حفظ إسم اللاعب و درجته كمتغيرات في ملف خارجي و عند بدء اللعبة يتم إستدعاء و قراءة الملف لتحميل المتغيرات: import pickle name = 'Test'; score = [100]; pickle.dump([name, score], open("trial.p", "wb")) في البرنامج أعلاه تم إستخدام pickle library لحفظ المتغيرين الإسم و الدرجة في ملف إسمه trial.p و لتحميلهم مرة أخرى يمكن أن نقوم بالاتي: name, score = pickle.load(open("trial.p","rb")) print(name, score) لاحظ أن عملية الكتابة تستخدم ب wb mode وهو الذي يمكننا من الكتابة في الملف، و عند الإستدعاء نستخدم rb mode وهو للقراءة من الملف الذي تم فيه حفظ القيم trial.p.
-
بما أنك تتعامل مع pandas إذن فإنه يمكنك تغيير عدد الأعمدة و الأسطر المعروضة كالتالي: import pandas as pd pd.options.display.max_columns = xxx pd.options.display.max_rows = xxx يمكنك تغيير xxx بالعدد المناسب. و إن كنت تتعامل مع Jupyter Notebook يمكنك أن تقوم بتحديد عرض السطر بإستخدام: import numpy as np np.set_printoptions(linewidth=160)
- 3 اجابة
-
- 1
-

-
لو تكرر ظهور الخطأ بعد تنفيذ ما وضحه @Ali Haidar Ahmad يمكنك التعامل مع نسخة tensorflow الأقدم و ذلك عن طريق الأسطر البرمجية التالية: import tensorflow.compat.v1 as tf tf.disable_v2_behavior()
- 2 اجابة
-
- 1
-

-
يمكنك ذلك عن طريق إستخدام الدالة loc كالتالي: import pandas as pd df = pd.DataFrame({'my_channel' : [100, 200, 500, 100, 50]}) df الناتج عبارة عن: my_channel 0 100 1 200 2 500 3 100 4 50 وبإستخدام الدالة loc الناتج يصبح: df.loc[(df.my_channel > 100),'my_channel']= 0 df my_channel 0 100 1 0 2 0 3 100 4 50
-
قد تكون المشكلة في إنشاء ال session التي يعمل عليها keras بالتالي يمكننا تنفيذ الأسطر البرمجية التالية: import tensorflow as tf sess = tf.compat.v1.Session(graph=tf.compat.v1.get_default_graph(), config=session_conf) tf.compat.v1.keras.backend.set_session(sess) و قم بتعريف tensorflow و keras في بداية البرنامج، و من ثم قم بإضافة Embedding لعملية الإستدعاء من layers: import tensorflow as tf from tensorflow import keras from tensorflow.keras.models import Sequential from tensorflow.keras.initializers import Constant from tensorflow.python.keras import backend as k from tensorflow.keras.layers import Flatten, Dropout, Dense,LSTM, Embedding model = Sequential() model.add(Embedding(1000, 128, input_length=512)) model.add(Flatten()) model.add(Dense(4, activation='softmax'))
-
DummyClassifier هو من أبسط الخوارزميات المستخدمة في عمليات التصنيف بإستخدام قواعد بسيطة، , ويعتبر من الجيد إستخدامه للمقارنة مع خوارزميات التصنيف الأخرى لا يحبذ إستخدامه كنموذج أساسي للتصنيف في حال البيانات الحقيقية، مثال: import numpy as np from sklearn.dummy import DummyClassifier X = np.array([-1, 1, 1, 1]) y = np.array([0, 1, 1, 1]) dummy_clf = DummyClassifier(strategy="most_frequent") dummy_clf.fit(X, y) print(dummy_clf.predict(X)) print(dummy_clf.score(X, y)) في هذه الحالة قمنا بإستخدام القيمة الأكثر تكراراً كإستراتيجية للتنبؤ. والان يمكننا التعامل مع مثال نقوم فيه بمقارنة نموذجين للتصنيف: from sklearn.model_selection import train_test_split from sklearn.neighbors import KNeighborsClassifier from sklearn.datasets import load_breast_cancer from sklearn.dummy import DummyClassifier Data = load_breast_cancer() X = Data.data y = Data.target X_train, X_test, y_train, y_test = train_test_split( X, y, test_size = 0.3, random_state = 0) test_scores = [] dclf = DummyClassifier(strategy = 'most_frequent', random_state = 0) dclf.fit(X_train, y_train) score = dclf.score(X_test, y_test) test_scores.append(score) clf = KNeighborsClassifier(n_neighbors = 5) clf.fit(X_train, y_train) score = clf.score(X_test, y_test) test_scores.append(score) test_scores في المثال السابق عرفنا قاعدة بيانات والتي تحتوي على 30 خاصية تستخدم لتصنيف الورم إلى سرطاني و غير سرطاني two class classification problem و قمنا بتعريف قيم المدخلات و المخرجات و من ثم تقسيم البيانات بنسبة 30% للإختبار و 70% للتدريب. و بعد ذلك قمنا بتعريف النموذجين، الأول هو التجريبي لمعرفة هل تصنيفنا يتم بصورة جيدة أم لا، و الثاني عبارة عن نموذج KNeighborsClassifier والذي يستخدم تصنيف أقرب 5 عينات للتنبؤ n_neighbors=5 و بمقارنة النتائج حصلت على: [0.631578947368421, 0.9473684210526315] للنموذجين على التوالي، بالتالي فإن إستخدام KNeighborsClassifier جيد جداً بمقارنة نتائجه مع DummyClassifier.
-
تعرف escape كالتالي: escape(string) Return string with all non-alphanumerics backslashed; this is useful if you want to match an arbitrary literal string that may have regular expression metacharacters in it. وبالتالي فهي ترجع القيم غير الرقمية و الحرفية في شكل string مسبوقة ب \. وبذلك تفيد في حال التعامل مع نصوص قد تحتوى على علامات خاصة. مثال: import re re.escape('^a.*$') # \\^a\\.\\*\\$ والناتج في هذه الحالة يكون: \\^a\\.\\*\\$ هو نفس القيمة المدخلة مسبوقة بعلامات ال backslash في حال الحروف الخاصة.
-
يمكنك ذلك عن طريق تنفيذ البرنامج التالي: import os print os.path.abspath(__file__) أو الأسطر البرمجية التالية والتي تعمل على MAC: import os file_path = os.path.realpath(__file__) كما يمكنك أيضاً إستخدام مكتبات sys, os: import sys, os print('sys.argv[0] =', sys.argv[0]) pathname = os.path.dirname(sys.argv[0]) print('path =', pathname) print('full path =', os.path.abspath(pathname))
-
يمكن إستخدامه لمعرفة التشابه و الإختلاف بين مجموعتين، و ذلك يتم عن طريق حساب ناتج الضرب النقطي للمجموعتين، راجع المثال: import numpy as np from sklearn.metrics.pairwise import cosine_similarity x = np.random.rand(1000,1000) y = np.random.rand(1000,1000) cosine_similarity(x, y) وناتج الدالة cosine_similarity في هذه الحالة عبارة عن مصفوفة ابعادها 1000*1000 لحاصل تشابه القيم في كل من x,y. يمكننا تنفيذ إستخدام CosineSimilarity في نموذج بيانات boston dataset والتي تعتبر من البيانات المستخدمة في عملية regression لتحديد سعر المنزل بإستخدام عدد من الخصائص. from keras import models, layers from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split X, y = load_boston(return_X_y=True) X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.4) print(X_train.shape, y_train.shape,X_test.shape, y_test.shape) model = models.Sequential() model.add(layers.Dense(13, activation='linear', input_shape=(13,))) model.add(layers.Dense(26, activation='sigmoid')) model.add(layers.Dense(1, activation='tanh')) model.compile(optimizer='rmsprop',loss=tf.keras.losses.CosineSimilarity(axis=1),metrics=['mse']) model.fit(X_train, y_train, epochs=3, batch_size=200) في هذه الحالة قمنا بإستخدام tf.keras.losses.CosineSimilarity و التي تعمل على مستوى الخصائص و ذلك عبر تحديد axis=1.
-
Mean Squared Error (MSE) من أشهر دوال حساب الخطأ في regression problems والتي تحسب متوسط مربعات الفروقات بين القيم المتوقعة من النموذج و القيم الحقيقية، تمثل رياضياً ب: والدالة توجد في sklearn.metrics ويمكن مناداتها كالتالي: from sklearn.metrics import mean_squared_error y_true = [3, -0.5, 2, 7] y_pred = [2.5, 0.0, 2, 8] mean_squared_error(y_true, y_pred) والان يمكننا أن نقوم بعرض مثال لطريقة تطبيق mse على بيانات boston dataset: from keras import models, layers from sklearn.datasets import load_boston from sklearn.model_selection import train_test_split X, y = load_boston(return_X_y=True) X_train, X_test, y_train, y_test = train_test_split( X, y, test_size=0.4) print(X_train.shape, y_train.shape,X_test.shape, y_test.shape) model = models.Sequential() model.add(layers.Dense(13, activation='linear', input_shape=(13,))) model.add(layers.Dense(26, activation='sigmoid')) model.add(layers.Dense(1, activation='tanh')) model.compile(optimizer='rmsprop',loss=tf.keras.losses.mean_squared_error,metrics=['mse']) model.fit(X_train, y_train, epochs=3, batch_size=200) لاحظ ناتج تنفيذ النموذج و الذي يكون عبارة عن 3 حلقات epochs في كل مرة يتم فيها تدريب النموذج على عدد 200 مدخل و يتم تقييم النموذج بإستخدام mean_squared_error.
-
Huber loss هي إحدى دوال حساب الخطأ في regression و تعتبر أقل تأثراً بالقيم الشاذة او المتطرفة من mean squared error. تعتبر Huber loss من الدوال التربيعية تستخدم mean squared error loss في حال كان الفرق بين القيم الحقيقية و المتوقعة صغير و تعتبر الدالة خطية و تحسب عن طريق mean absolute error في حال كان الفرق كبير، المتغير دلتا هو من يحدد نقطة التحول من الحساب الخطي للحساب التربيعي في الدالة، لاحظ المعادلة: لاحظ المثال التالي: from tensorflow import keras yActual = [4, -1.5, 5, 2] yPredicted = [3.5, 1, 5, 3] huberObject = keras.losses.Huber(delta=0.5) huberTensor = huberObject(yActual, yPredicted) huber = huberTensor.numpy() print(huber) والناتج هو قيمة 0.40625 والتي نتجت من العمليات الحسابية التالية:
-
Dense هي أول طبقة لدينا، لذلك يجب أن نمرر شكل البيانات التي يجب أن تستقبلها في هذه الحالة، في حالة عدم تحديد الشكل تقوم الطبقة بأخذ شكل البيانات من الطبقة السابقة وفي هذه الحالة Dense كانت الطبقة الأولى بالتالي نمرر شكل البيانات. لاحظ أنك قمت بتعريف maxlen وهو متغير ال padding الذي يقوم بتحويل شكل البيانات المدخلة من 25000, 25000 لكل من ال input_train, input_test على التوالي ل (25000, 20) لكل منهما. بالتالي نمرر maxlen لمتغير input_length في طبقة Embedding ويصبح البرنامج: from keras.layers import Dense from keras.datasets import imdb from keras.models import Sequential from keras.preprocessing import sequence from keras.layers import Embedding, SimpleRNN,Flatten,Dense maxlen = 20 (input_train, y_train), (input_test, y_test) = imdb.load_data(num_words=10000) print(len(input_train), 'train sequences') print(len(input_test), 'test sequences') print('train_shape',input_train.shape) print('test_shape',input_test.shape) print('Pad sequences (samples x time)') print(input_train[0]) input_train = sequence.pad_sequences(input_train, maxlen=maxlen) input_test = sequence.pad_sequences(input_test, maxlen=maxlen) print('input_train shape:', input_train.shape) print('input_test shape:', input_test.shape) print(input_train[0]) model = Sequential() model.add(Embedding(10000, 16,input_length=maxlen)) model.add(Flatten()) model.add(Dense(32, activation='relu')) model.add(Dense(1, activation='sigmoid')) model.compile(optimizer='rmsprop', loss='binary_crossentropy', metrics=['acc']) history = model.fit(input_train, y_train, epochs=2, batch_size=32, validation_split=0.2)
-
Logcosh: هي إحدى دوال حساب الخطأ في keras والتي تستخدم لوغريثم جيب التمام في حساب الفرق بين القيم الحقيقية و المتوقعة من النموذج. تكون على صيغة: tf.keras.losses.log_cosh(y_true, y_pred) حيث ان tf ترمز ل tensorflow و y_true للقيم الحقيقية، y_pred للقيم المتوقعة من النموذج. مثال: from tensorflow import keras yActual = [4, -1.5, 5, 2] yPredicted = [3.5, 1, 5, 3] logcoshObject = keras.losses.LogCosh() logcoshTensor = logcoshObject(yActual, yPredicted) logcosh = logcoshTensor.numpy() print(logcosh) والناتج عبارة عن 0.59186584 وهي حاصل (x ** 2) / 2 لأن القيم صغيرة و يتم حسابه ك abs(x) - log(2) في حال كانت القيم كبيرة، ذلك يعنى انه مثل mean_squared_error و لكنه أقل تأثر بالقيم المتوقعة بصورة خاطئة.
-
KL-Divergence هي طريقة لحساب نسبة الفقد او الخطأ بين البيانات الحقيقية و الناتجة من النموذج، والتي تعتمد في القياس على Kullback-Leibler divergence والتي تقوم بحساب الخطأ بالطريقة التالية: loss = y_true * log(y_true / y_pred) يتم تعريف KL-Divergence في tensorflow بالطريقة التالية: tf.keras.losses.KLDivergence( reduction=losses_utils.ReductionV2.AUTO, name='kl_divergence' ) مثال لطريقة تطبيق الKL-Divergence: import tensorflow as tf y_true = [[0, 1], [0, 0]] y_pred = [[0.6, 0.4], [0.4, 0.6]] kl = tf.keras.losses.KLDivergence() kl(y_true, y_pred).numpy() والناتج عبارة عن 0.45814306 وهي قيمة الخطأ بين y_true و y_pred والتي يتم حسابها بالعلاقة في الشرح أعلاه.
-
يتم إنشاء بيانات بصورة عشوائية بإستخدام دالة make_regression المتوفرة في مكتبة sklearn.datasets. من أهم التعريفات التي يتم تمريرها للدالة هي عدد العينات المراد الحصول عليها، عدد الخصائص features و يمكن أيضاً تمرير n_informative والتي تشير لعدد الخصائص ذات التأثير على قيم y في النموذج. في المثال التالي يتم إنتاج 200 عينة عشوائية، بإستخدام 5 خصائص بها 2 من informative features: import pandas as pd import seaborn as sns import numpy as np from sklearn import datasets import matplotlib.pyplot as plt # إنشاء البيانات X, y = datasets.make_regression(n_samples=200, n_features=5, n_informative=2) # إنشاء بانداس لنقوم بحساب العلاقات بين الخصائص df = pd.DataFrame(X) df.columns = ['feature1', 'feature2', 'feature3', 'feature4', 'feature5'] df['target'] = y corr = df.corr() #heat_map رسم نسبة قوة العلاقة بين الخصائص بإستخدام f, ax = plt.subplots(figsize=(9, 6)) mask = np.triu(np.ones_like(corr, dtype=bool)) cmap = sns.diverging_palette(230, 20, as_cmap=True) sns.heatmap(corr, annot=True, mask = mask, cmap=cmap)